Unidade VIII - Biologia Molecular
2. Bibliotecas de DNA
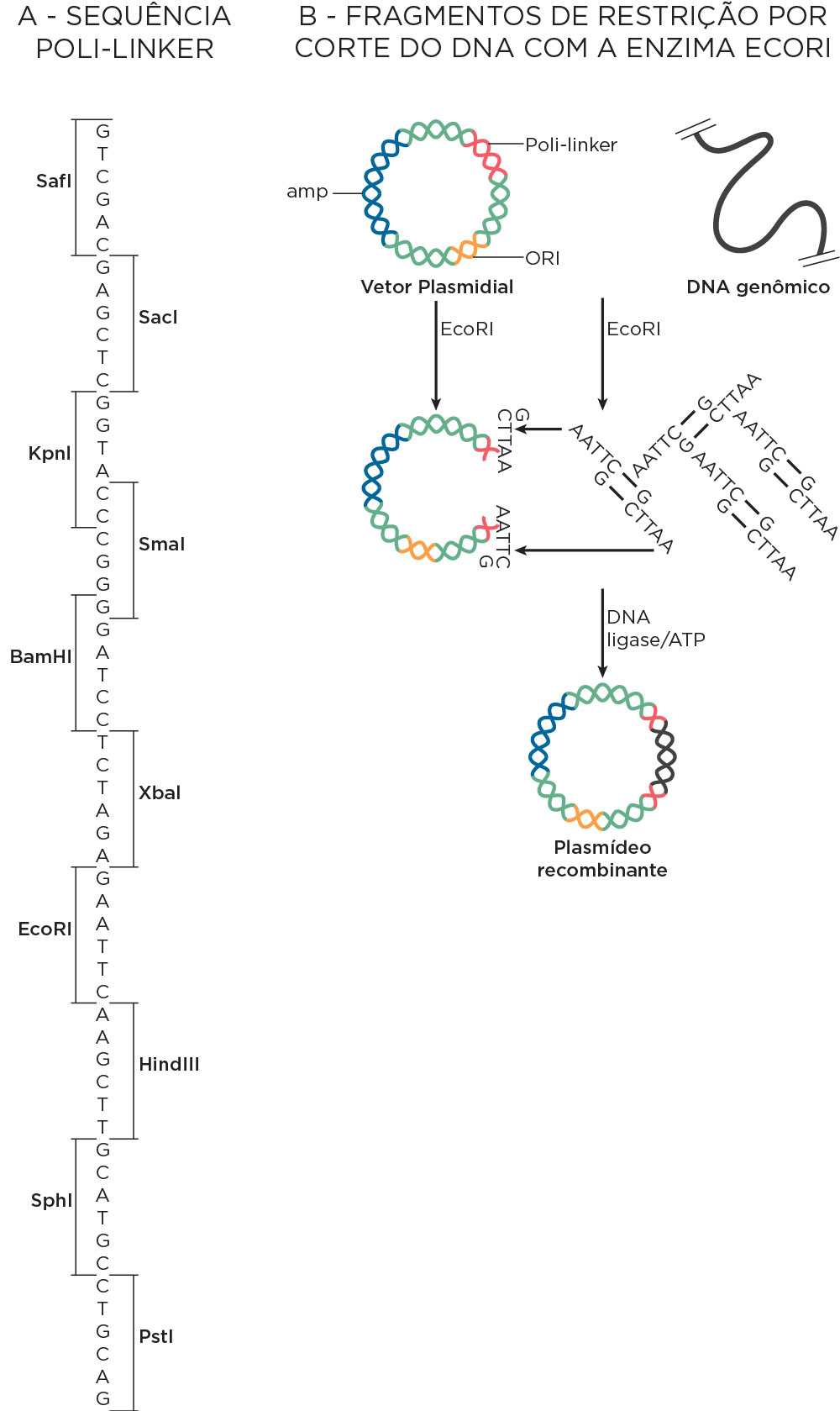
Os cromossomos inteiros (biblioteca genômica) ou em parte (biblioteca gênica) são clonados para isolar o DNA de uma célula, organismo, ou fragmentos de tecidos, por meio da digestão com enzimas de restrição de ambos, vetor e inserto com a mesma enzima. Após formação do DNA recombinante pela ligase, este é inserido nas células hospedeiras por meio da transformação e após vários ciclos de replicação são formados as colônias de clones de bactérias contendo o inserto.
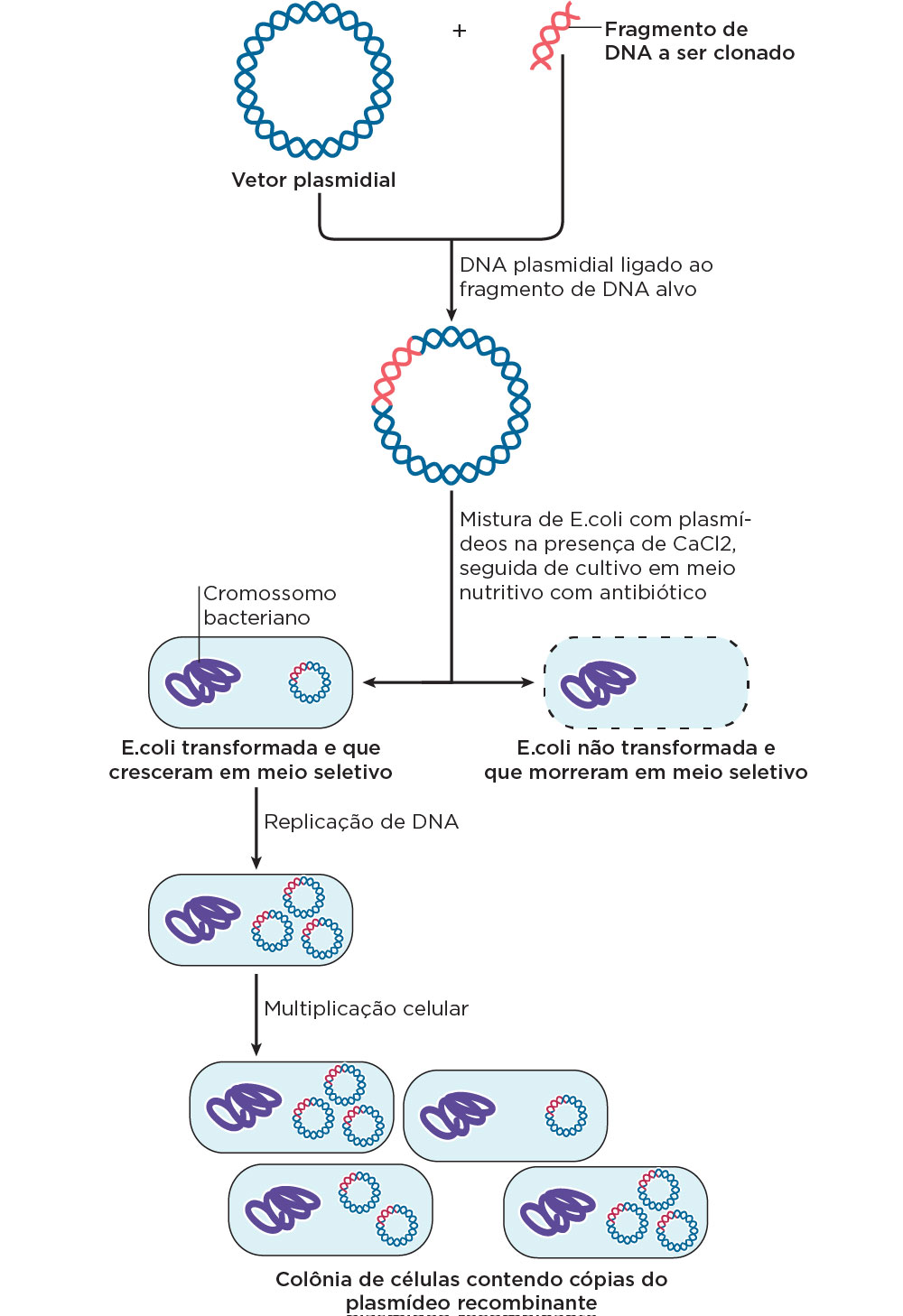
Um dos métodos de transformação do hospedeiro pela inserção do plasmídeo exógeno é a utilização de reagentes químicos. O tratamento da membrana com um sal, geralmente o cloreto de cálcio, faz com que a membrana de E.coli fique permeável ao DNA, no momento em que o plasmídeo entra na célula . Além da transformação química, é possível realizar a permeabilização da membrana, por meio de choques, provocados por um aparelho eletroporador.
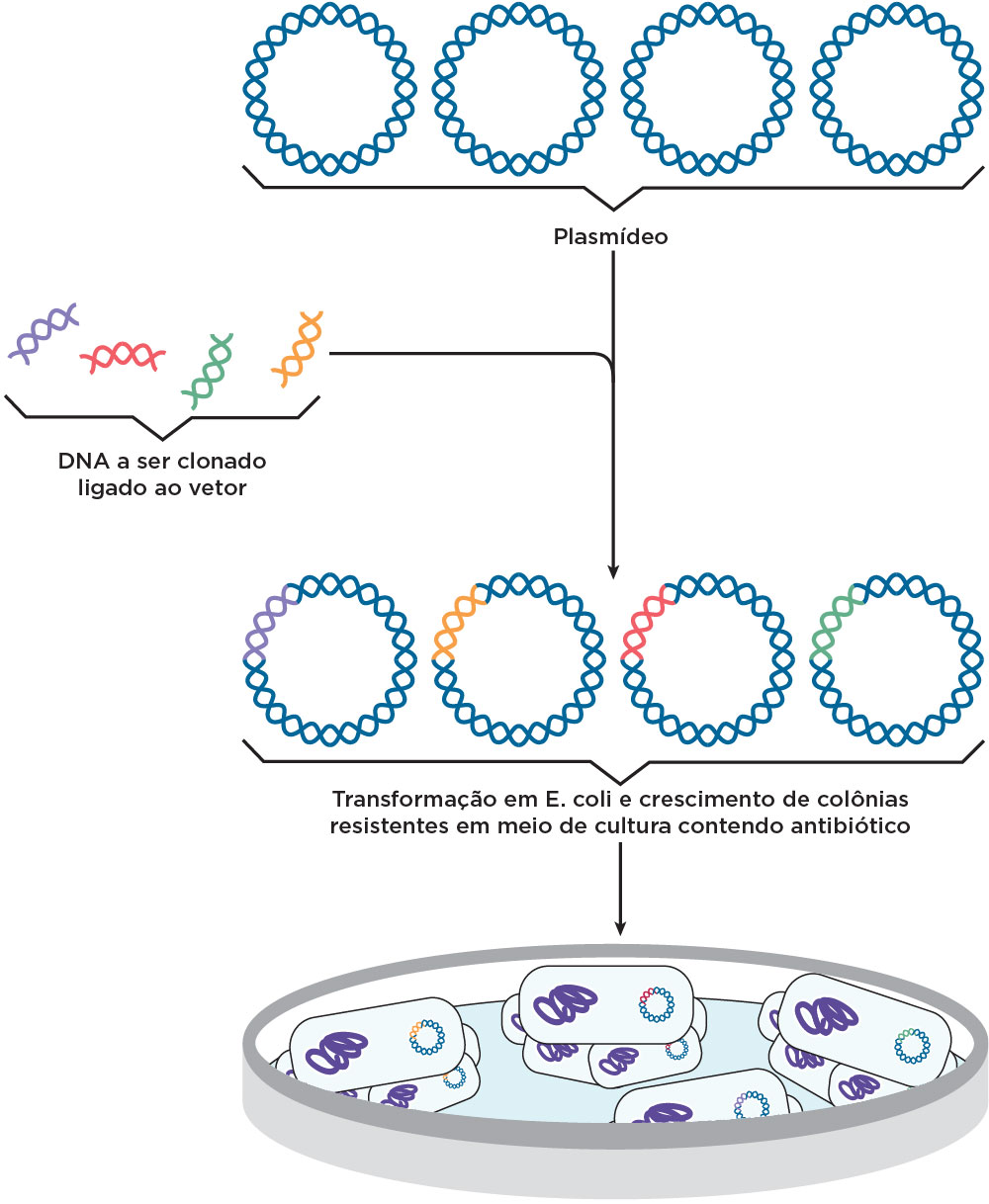
Como dito anteriormente, após a transformação, a seleção poderá ser realizada utilizando um antibiótico e os marcadores fenotípicos para a cor. As bactérias transformadas com plasmídeo serão resistentes ao antibiótico utilizado, enquanto as demais, que não estão transformadas com o plasmídeo, morrerão. A seleção fenotípica pela cor é realizada pela adição de um substrato para a beta-galactosidase no meio de crescimento das bactérias. Num processo normal, sem inserto, o IPTG (isopropil-B-tio-galactosídeo) após ser clivado pela beta-galactosidase leva à formação de uma cor azul nas colônias, isso porque o sítio para a produção da enzima beta-galactosidase (gene lacZ), contendo o sítio SMC, é capaz de produzir a enzima. Contudo, após a ligação do inserto no SMC, o gene lacZ é interrompido e o seu produto protéico não será produzido, resultando em colônias brancas .
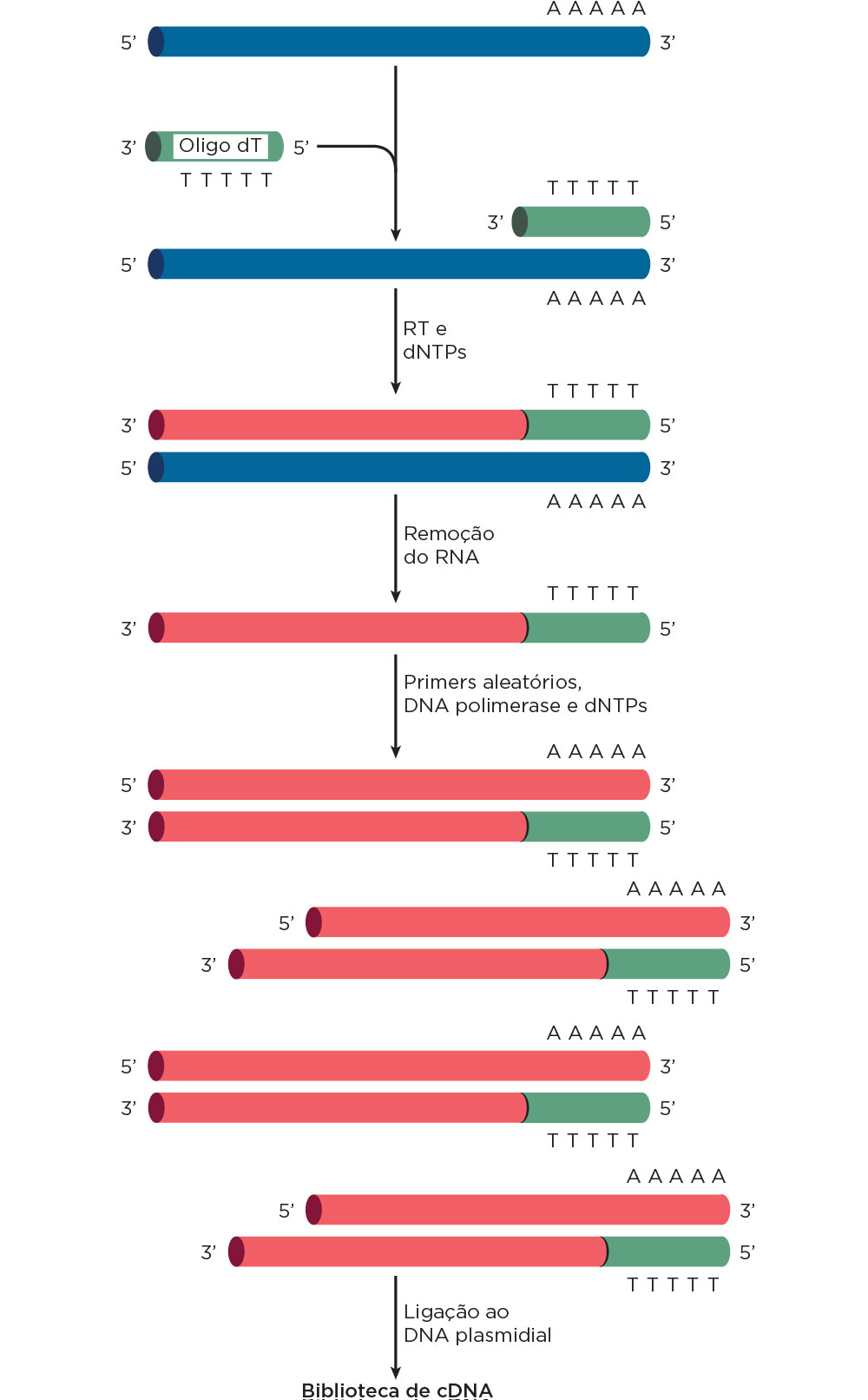
A biblioteca gênica consiste na clonagem de um gene ou segmento gênico específico por meio da extração de RNA, seguida da sua conversão em DNA complementar ao RNA (cDNA), pela enzima Transcriptase Reversa (RT extraída de vírus). O processo envolve a utilização de um primer, por exemplo, poliT (liga à cauda poliA do RNAm) para síntese do cDNA pela RT. O cDNA é convertido em DNA dupla fita (dsDNA) pelo uso da enzima DNA polimerase e PCR descrita a seguir, e então inserido num vetor de clonagem após digestão do produto com a mesma enzima de restrição utilizada na clivagem do vetor e ligado pela DNA ligase, ou pela utilização de kits. O processo seguinte é o mesmo descrito anteriormente .