Unidade VIII - Biologia Molecular
1. Técnicas de DNA recombinante
Corresponde ao mecanismo de isolar e caracterizar qualquer gene ou sequências de DNA de qualquer organismo. A engenharia genética, seguida de clonagem, são métodos utilizados na tecnologia do DNA recombinante, para a construção de Bibliotecas Gênicas e Genômicas. A engenharia genética realiza a manipulação direta de ácidos nucleicos (DNA, RNA) com o objetivo de transferir uma informação genética de um organismo a outro.
Clonagem
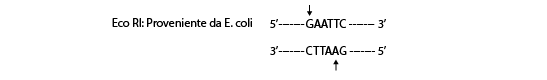
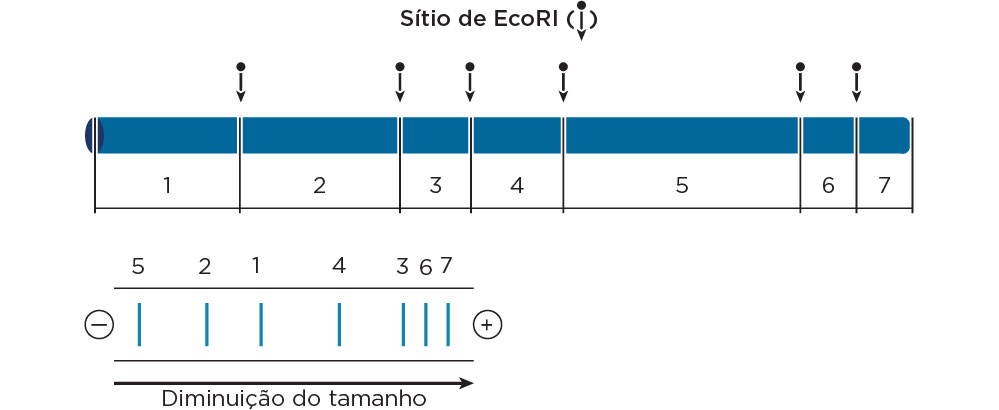
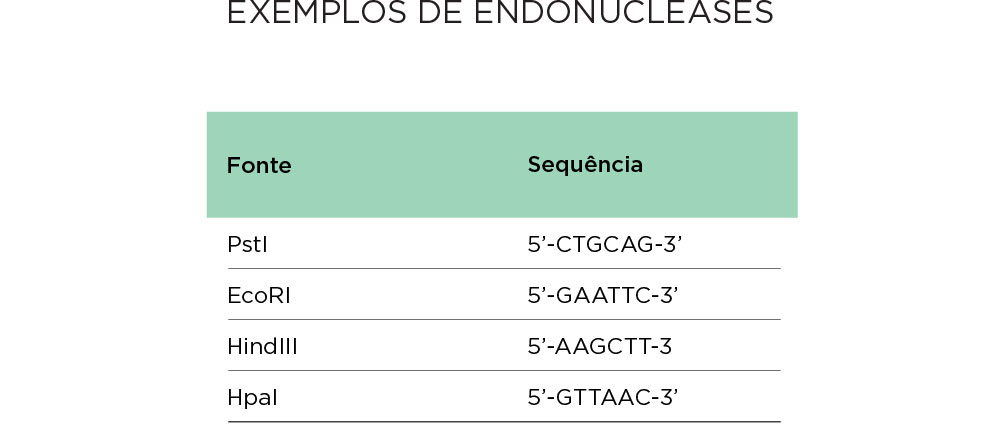
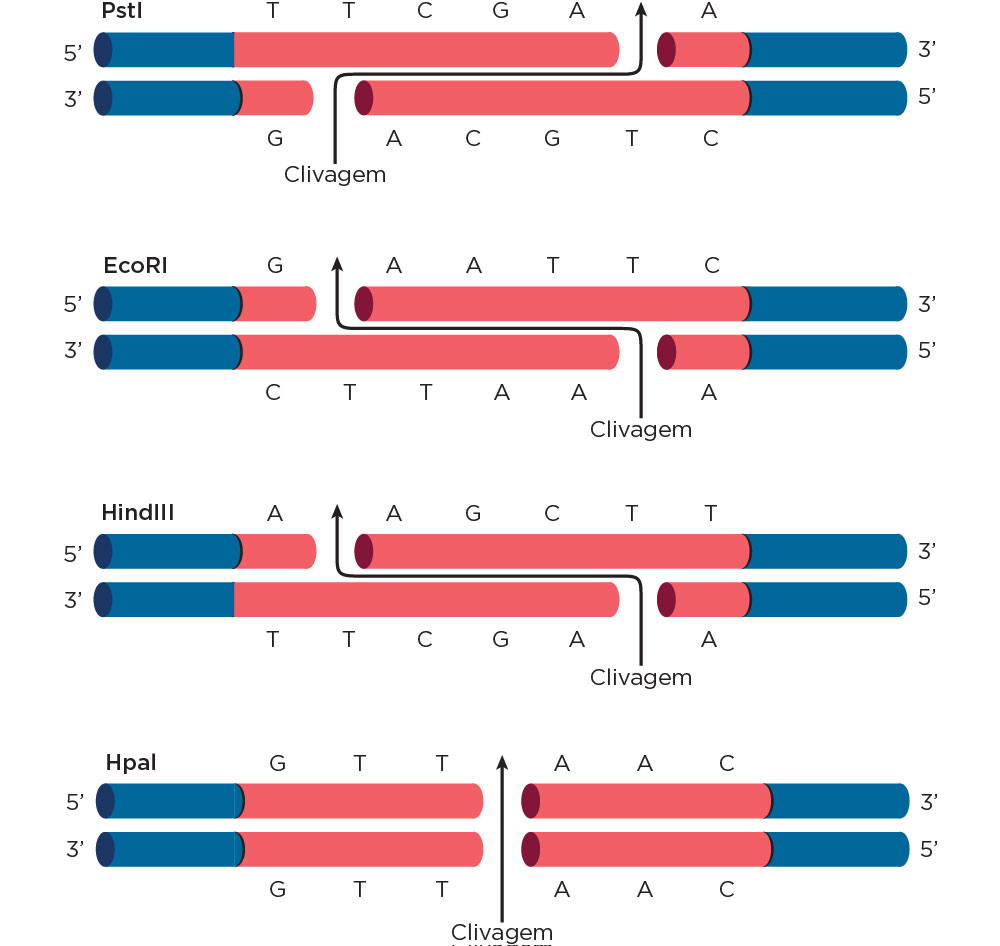
Um segmento de DNA é ligado a um pequeno cromossomo autoreplicante e inserido em uma célula hospedeira apropriada. As endonucleases de restrição, que são utilizadas nesse processo, correspondem a enzimas que fazem cortes internos nas moléculas de DNA de forma sítio específicas, sendo designadas pelo uso da primeira letra do gênero do micro-organismo que a produz e as duas primeiras letras da espécie. O número que a acompanha corresponde à ordem de identificação. Ex.: EcoR1 - E = Escherichia ; co = coli, 1 = primeira identificada.
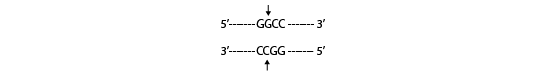
Existem aproximadamente 400 enzimas de restrição diferentes, cuja função é de proteção do material genético da bactéria contra DNAs exógenos, como os DNAs virais. Atuam por reconhecer sequências palindrômicas no DNA, ou seja, em que a leitura é a mesma em ambos os sentidos na fita dupla. Os cortes gerados podem ser lisos e coesivos. As extremidades coesivas geram pontas unifilamentares complementares podendo ser reunidas com outros DNAs cortados com a mesma enzima. No caso das enzimas que geram extremidades lisas, é necessária a ligação de sequências denominadas de adaptadores (linkers). A ligação fosfodiéster é realizada pela enzima DNA ligase.
5’...→GAATTC...3’
3’... CTTAAG←...5
Coesivas:
Lisas:
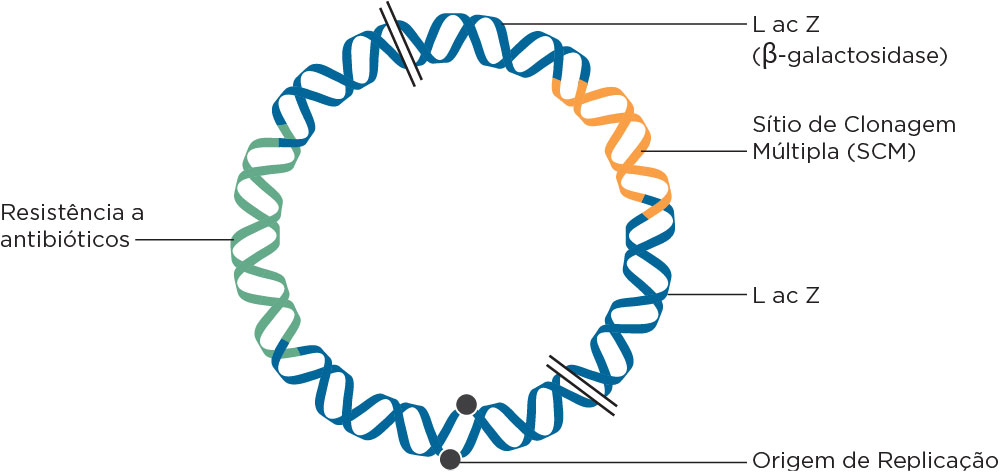
Nesse processo são também utilizados os vetores de clonagem que são moléculas de DNA capazes de amplificar, em centenas de cópias, a informação genética que neles foi inserida. O mais comum na rotina laboratorial é o plasmídeo . A molécula de DNA de um plasmídeo é circular, extracromossômico e bifilamentar pequeno (cerca de 3 mil pares de bases) encontrado em procariotos, que apresenta replicação independente do genoma desses organismos (autorreplicação) devido à presença do sítio OriC, além de possuir sequências para marcadores fenotípicos como para resistência à antibióticos (AmpR - Resistência à Ampicilina) e para expressão da beta-galactosidase. O DNA plasmidial também contém sítios para clivagem com várias endonucleases de restrição (ER) presentes uma única vez no genoma plasmidial, denominado sítio de clonagem múltipla (SMC), de policlonagem ou polilinker. Assim, a seleção de E.coli contendo o plasmídeo com o fragmento de interesse (inserto) é feita pela análise de sobrevivência em meio contendo antibiótico e pela não expressão da beta-galactosidase, uma vez que o sítio de clonagem está dentro do sítio LacZ, que será interrompido pelo inserto (mutação). O primeiro tipo de plasmídeo descrito foi o pSC101 conferindo resistência à tetraciclina, sendo que plasmídeos mais versáteis foram posteriormente desenvolvidos.