Unidade V - Regulação da expressão gênica em procariotos
As células de todo organismo, sejam uni ou pluricelulares, apresentam genes que possuem níveis constantes de expressão, chamados de constitutivos ou genes “de economia doméstica” (housekeeping) e seus produtos são constantemente requeridos pela célula, os quais permanecem “ligados”. Existem os genes cuja expressão é variável e estão sob influência de sinais moleculares específicos, tais como, fatores de crescimento, hormônios, e outros para que seus produtos sejam produzidos. Se todos os seus genes estivessem “ligados“ a todo tempo na célula, haveria um desperdício de proteínas que não estariam sendo utilizadas o tempo todo. O mecanismo de “ligar” e “desligar” genes é conhecido como Regulação da Expressão Gênica e é de importância vital para melhor utilização da energia disponível na célula, dado o alto custo da síntese protéica.
EXPRESSÃO GÊNICA EM PROCARIOTOS,
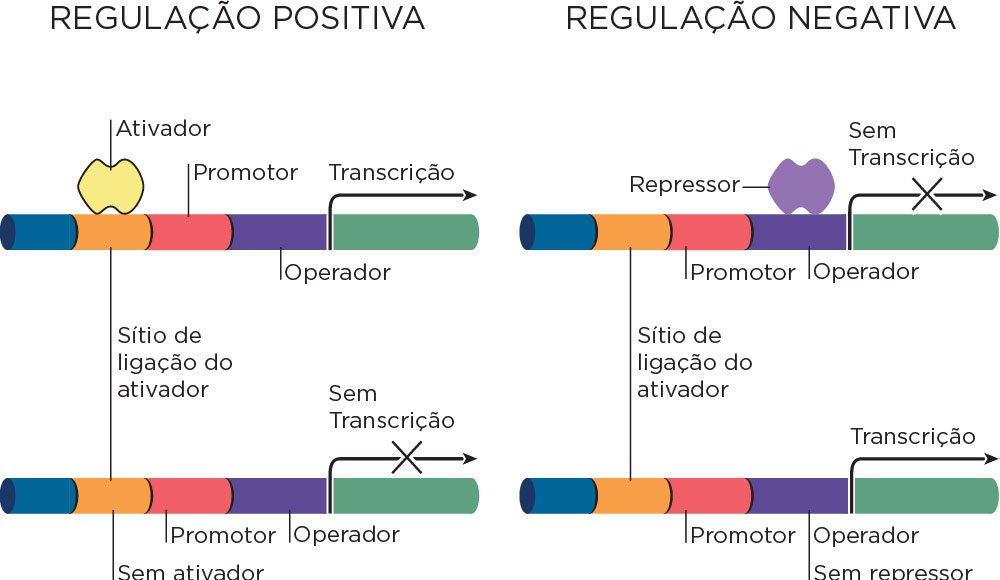
O termo expressão gênica refere-se ao processo em que a informação codificada por um determinado gene é funcional na forma de RNA ou será decodificada em uma proteína. Teoricamente, em qualquer uma das etapas do fluxo da informação genética, que vai do DNA à proteína pode levar a uma expressão gênica diferencial. Os genes podem ser regulados por sinais intrínsecos às celulas, bem como por sinais extracelulares. Para as bactérias, cujo crescimento pode ser realizado em meios de cultura específicos, esses sinais externos estão disponíveis no meio, e podem desencadear uma série de eventos controlados por ligação a proteínas reguladoras.Essas proteína podem ser reguladoras positivas da transcrição (ativadores) ou reguladoras negativas (repressores).
Por meio de ligação ao DNA em sítios específicos, as proteínas reguladoras podem assim controlar a expressão de genes específicos, aumentando, bloqueando ou reduzindo a síntese de RNA, sendo que um ativador aumenta as taxas de transcrição do gene que controla, enquanto os repressores reduzem ou anulam a transcrição gênica.
Nas bactérias, o controle da expressão gênica serve, principalmente, para permitir que as células se ajustem às mudanças nutricionais no ambiente, de forma que seu crescimento e divisão sejam otimizados. Muitos genes estão em complexos chamados operons; e nestes complexos, normalmente, existe uma proteína repressora e uma ativadora da transcrição de vários genes que, atuando em "parceria", determinam certas características.
OPERON LAC
O Operon Lac foi descoberto por François Jacob e Jaques Monod, em 1960, como um mecanismo responsável pela codificação de enzimas, que absorve e digere a lactose do meio. O Operon Lac consiste de um operador, um promotor e três genes estruturais. O RNAm do operon é chamado de policistrônico ou poligênico, pois possui informação para codificar as três proteínas, cujos genes beta-galactosidase, permease e transacetilase são transcritos em um único filamento de RNAm.
Assim, no metabolismo da lactose por E. coli a primeira enzima produzida pelo operon é a beta-galactosidade, uma enzima capaz de clivar a lactose em glicose e galactose, que assim servirão como fonte de carbono para a célula .
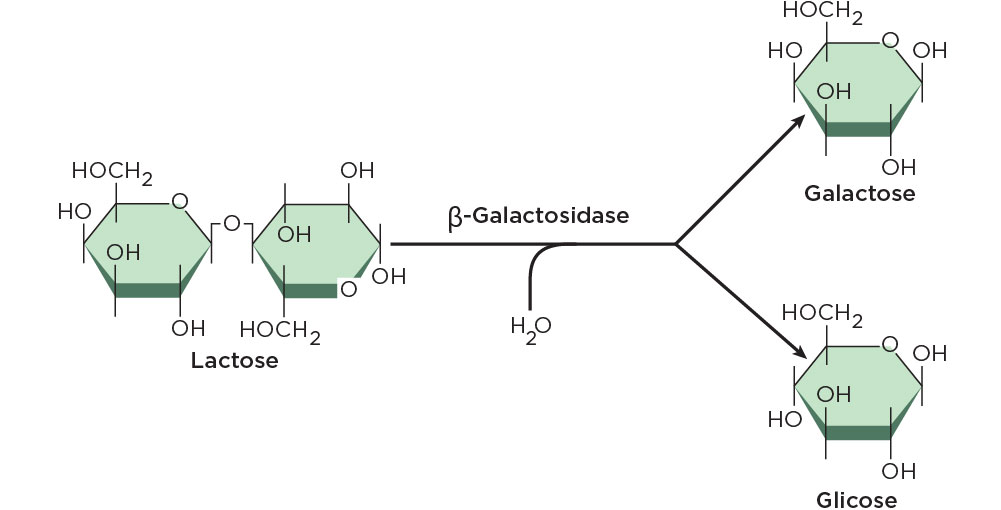
A beta-galactosidase é uma enzima indutível, ou seja, sua expressão varia de acordo com as necessidades celulares e, caso a bactéria esteja crescendo em meio rico em lactose, sua expressão será alta; no entanto, caso a fonte de carbono seja outro carboidrato, sua expressão será reduzida. A outra enzima do operon é a permease, que, como seu próprio nome indica, é a enzima responsável pelo transporte de lactose do meio extracelular para o meio intracelular através da membrana da célula bacteriana, pois a lactose, como a maioria dos carboidratos, não é capaz de atravessar a bicamada lipídica sem uma proteína carreadora.
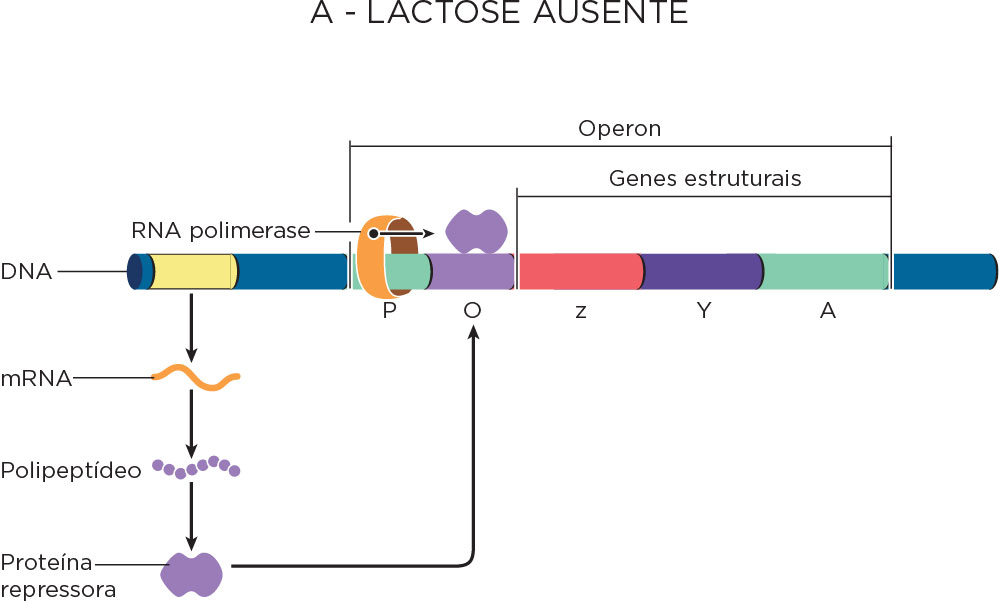
A última enzima, a tiogalactosídeo transacetilase tem função de inativar galactosídeos tóxicos, que também acabam sendo transportados para dentro da célula. A expressão dessas três proteínas é regulada por um outro gene (gene I) que codifica para um regulador Lac. O gene I é capaz de codificar um repressor que está faltando ou está inativado nos organismos com o gene I-. A região promotora (P), como visto anteriormente, é o local onde a RNA Polimerase se liga para começar a transcrição. Num operon, além da região promotora existe também o operador (O), os quais formam os locais de controle do operon. Na presença do produto do gene I, o operon é incapaz de ser codificado, pois o repressor está ligado ao operador , no entanto, caso o indutor, a própria lactose esteja presente, essa se liga ao repressor o qual não se liga ao DNA, não bloqueando a síntese das proteínas envolvidas no metabolismo da lactose .
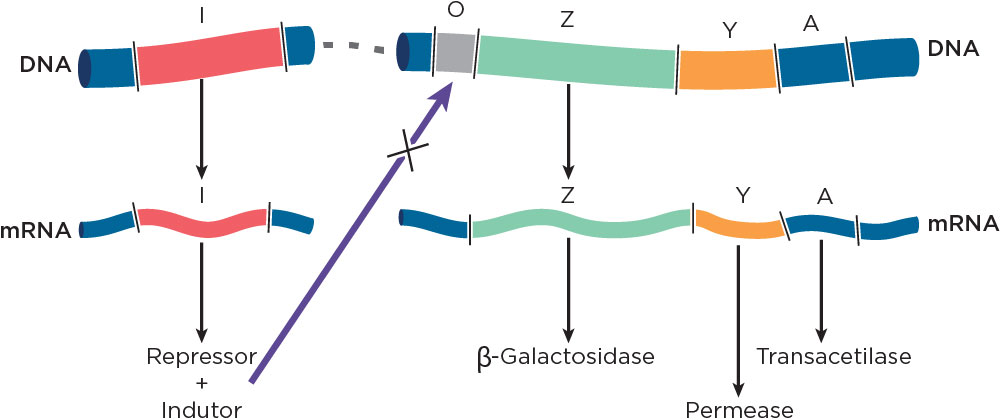

O isopropil-b-D-tiogalactosídeo (IPTG) é um indutor artificial do operon lac. Foi utilizando este composto que se descobriu o repressor lac codificado pelo gene I, que na ausência de um indutor liga-se ao operador e bloqueia a transcrição.
Relação Glicose e AMPc
Se a glicose estiver no meio de cultura da E. coli, ela será preferencialmente usada como fonte de energia, isto é, enzimas utilizadas no metabolismo dos outros carboidratos serão pouco ou nada expressos. Os efeitos negativos da glicose sobre os níveis de muitas enzimas são chamados de repressão catabólica. Esse efeito é moderado pela ação da proteína CAP (proteína ativadora de catabólitos), também chamada de CRP (proteína receptora de AMPc) e o metabólito celular AMPc (AMP cíclico). Em bactérias, o AMPc liga-se ao CAP, sendo que esse complexo CAP-AMPc é capaz de estimular a transcrição por se ligarem a determinados promotores. No operon lac, CAP se liga em regiões adjacentes ao promotor em que se liga a RNA polimerase, potencializando as taxas de transcrição.
A concentração de AMPc é controlada indiretamente pela concentração de glicose no meio. Foi observado que quando os níveis de glicose estão altos, os níveis de AMPc estão baixos e não é capaz de se ligar ao DNA. Quando são adicionados AMPc exógeno ao meio, a célula é capaz de metabolizar outros açúcares, como a arabinose e a lactose. É interessante notar que o AMPc em procariotos tem função semelhante ao AMPc em eucariotos. Nesses, quando os níveis de glicose circulante estão baixos, há o estímulo para a produção de glucagon (“hormônio da fome”), o que leva a aumentar os níveis de AMPc dentro das células sensíveis a hormônios.
A ligação da CAP tem dois efeitos, cria uma curvatura no DNA e reforça a fraca ligação entre polimerase e promotor , sendo que ambos os efeitos são importantes para o aumento nas taxas de transcrição. Dessa forma, quando os níveis de glicose estão baixos, a concentração do AMPc é alta e o complexo CAP-AMPc se liga ao DNA melhorando a síntese pelo operon lac .
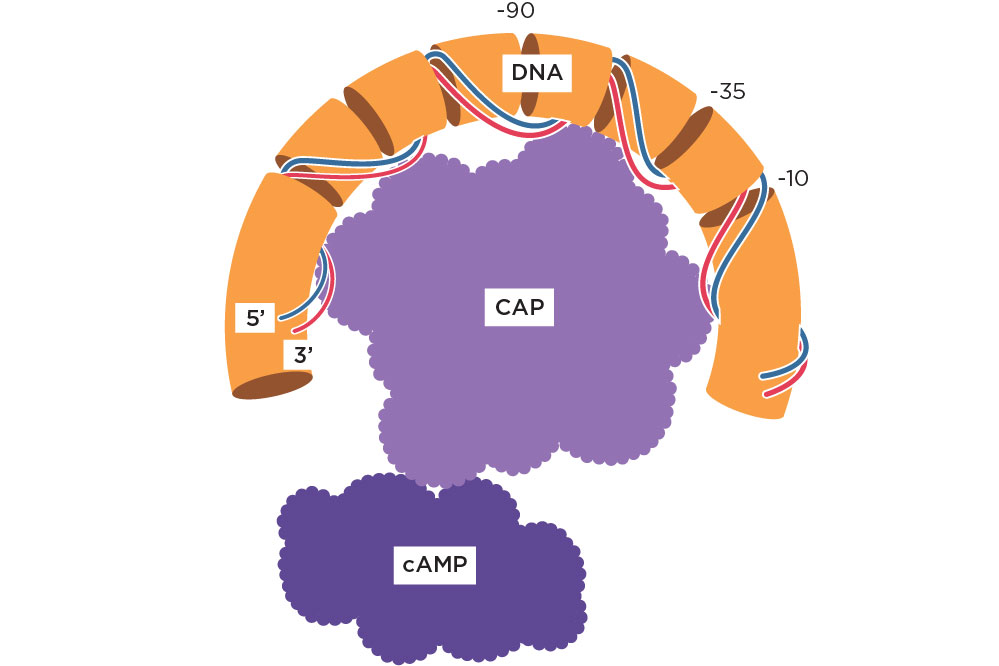
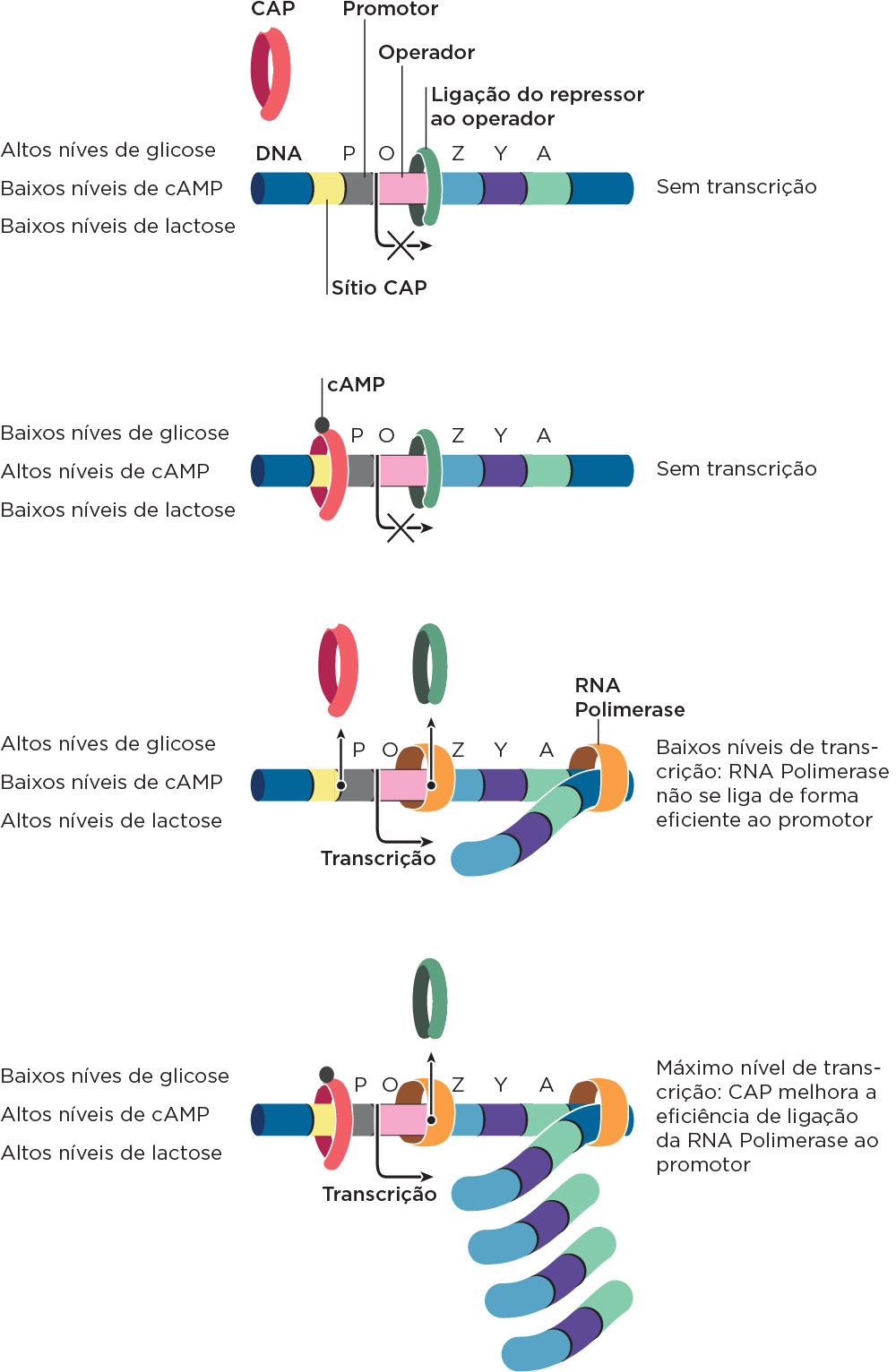
Os operons podem ser apresentar com regulação positiva e/ou com regulação negativa. Um operon está sobre controle regulatório positivo quando os genes são expressos (transcritos) na presença de uma proteína que se liga ao operador e sobre negativo, quando os genes são expressos na ausência de uma proteína repressora ligada ao operador. O operon lac apresenta os dois modelos regulatórios, sendo o primeiro representado pela ligação do AMPc-CAP potencializando a transcrição e o segundo representado pela proteína repressora ligada à lactose, a qual se desligará do operador permitindo a ação da RNA polimerase na transcrição do genes Z, Y e A .
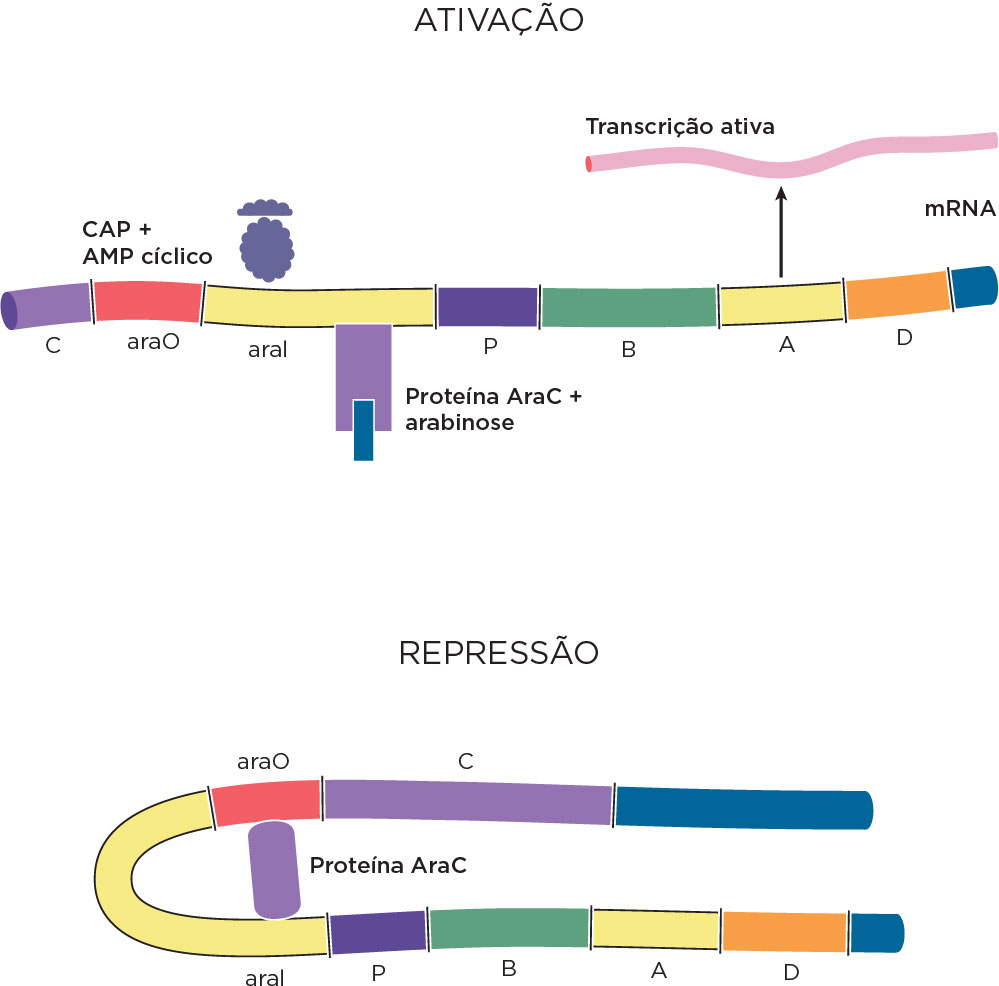
OPERON ARA
Assim como a lactose, a arabinose não é utilizada diretamente como fonte de carbono, ou seja, ela precisará ser modificada a xilulose-5-fosfato para ser utilizada no metabolismo. Os genes araBAD não são expressos na ausência de arabinose. O operon arabinose (ara) codifica para três enzimas necessárias para converter arabinose em xilulose 5-fosfato, um produto intermediário da via pentose fosfato. O operon da arabinose é formado por três genes estruturais que codificam:
- araA: arabinose isomerase (converte arabinose em ribulose)
- araB: ribulocinase (fosforila ribulose)
- araD: ribulose-5-fosfato epimerase (converte ribulose-5-fosfato a xilulose-5-fosfato a qual pode ser metabolizada pela via das pentoses)
O operon ara também está sobre controle duplo, sendo que é preciso a presença do complexo CAP-AMPc e a ligação da arabinose à proteína C para que a transcrição seja eficiente. Além do gene regulador ara C, que codifica a proteína C, existe uma região controladora com operador (O) e outra reguladora (ara I). No operon ara, a proteína C apresenta regulação positiva, atuando como ativador, e negativa, inibindo o araBAD, dependendo das condições celulares. O fator da chave liga/desliga para esse operon é a própria arabinose. Diferente do Operon Lac, na presença de arabinose, esta se ligará à proteína C a qual se ligará à região I do DNA, permitindo a transcrição gênica também na presença do complexo CAP-AMPc. Na ausência de arabinose, a proteína C também se liga à região I do DNA, fazendo com que ocorra uma dobra (alça) na molécula para que a mesma proteína C interaja com o operador O, não permitindo que a RNA polimerase inicie a transcrição do Operon Ara .
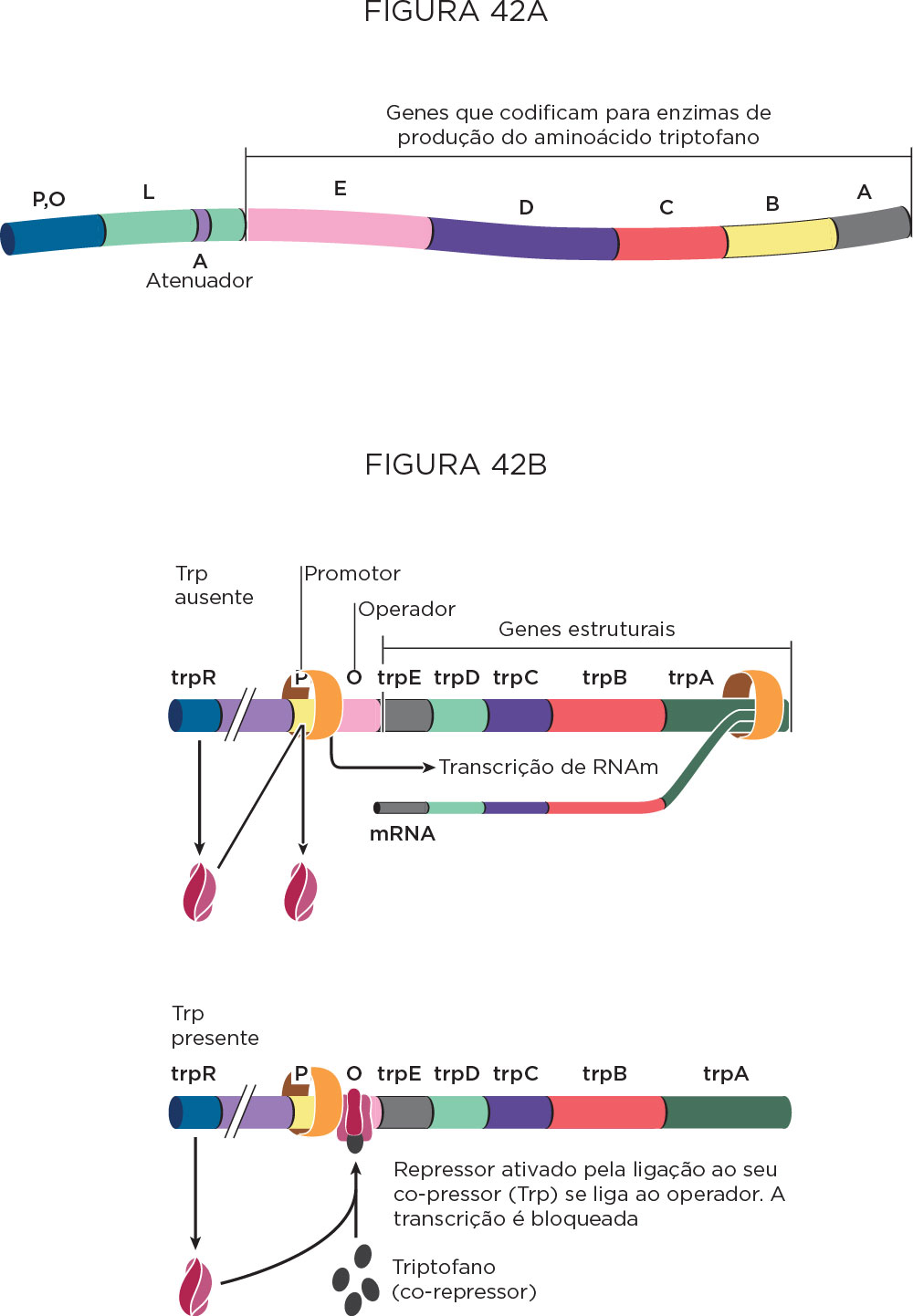
OPERON TRP
No Operon Trp, diferente dos dois tipos de regulação apresentados anteriormente, a ligação do triptofano torna o repressor ativo para controlar a síntese das cinco enzimas envolvidas na biossíntese do triptofano a partir do corismato. Para melhor entender esse modelo de operon é importante lembrar que em procariotos a transcrição e a tradução são processos acoplados, sendo que a tradução começa antes do término da transcrição. Os genes estruturais E, D, C, B, A codificam as enzimas que atuam na biossíntese do aminoácido triptofano. Além desses genes, a sequência líder, localizada imediatamente antes do gene E, atuará de forma a regular a transcrição das cinco enzimas. Como os demais, esse operon apresenta a sequência promotora seguida da sequência operadora .
O repressor trp codificado pelo gene trp é uma proteína alostérica de ligação ao DNA. No controle alostérico ocorre alteração na estrutura terciária ou quaternária de uma enzima induzida pela ação de uma molécula ligante, que pode ser um ativador, um inibidor, um substrato, ou os três. Quando não há triptofano, o repressor está em uma conformação que é incapaz de se ligar ao DNA, no entanto, quando o repressor se liga de forma reversível ao triptofano, este adquire uma conformação que faz com que se liga firmemente ao sítio operador. Desse modo, os níveis de triptofano na célula podem controlar a transcrição de genes necessários para sua própria síntese:
- ↓ [triptofano]: proteína repressora em conformação incapaz de se ligar ao DNA.
- ↑ [triptofano]: ligação do triptofan à proteína repressora, que será capaz de se ligar ao sítio operador no DNA pela mudança conformacional após ligação ao ligante, levando à redução da síntese dos genes estruturais.
Assim, o triptofano controla a sua própria síntese. Se há excesso de triptofano, este se liga ao repressor, o qual adquire uma mudança conformacional ligando-se ao operador. O operador se superpõe ao promotor para o início de transcrição, ou seja, o repressor, que está ligado ao operador, impede que a RNA polimerase se ligue eficientemente ao promotor trp impedindo a transcrição dos genes estruturais envolvidos na biossíntese do aminoácido triptofano.
Regulação por atenuação: importância da sequência líder
Uma característica importante dos procariontes é a ausência de compartimentalização nas células, ou seja, não existe separação das estruturas celulares por membranas. Sendo assim, o DNA procariótico está solto no citoplasma permitindo que os processos de transcrição e tradução ocorram simultaneamente. A atenuação faz uso desse rígido acoplamento entre esses dois processos para controlar a expressão gênica.
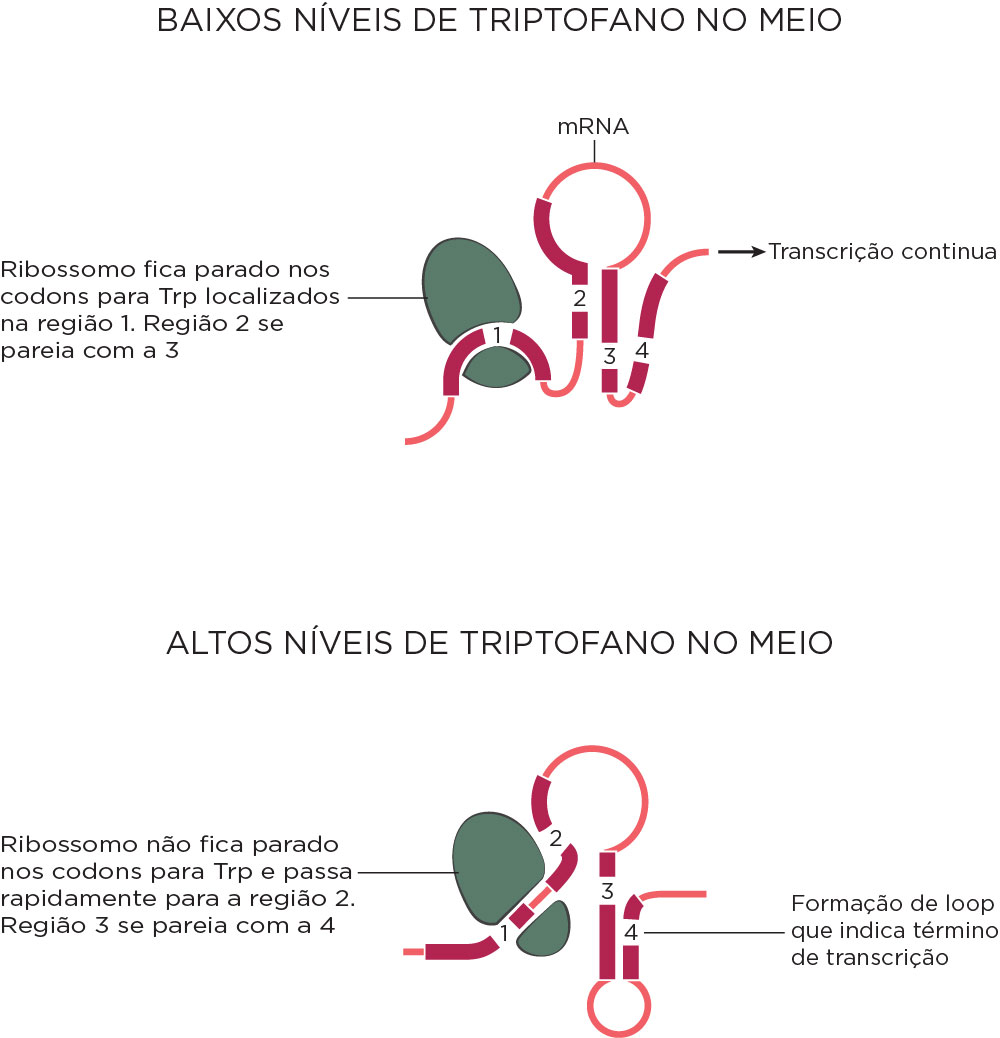
A regulação de uma terminação prematura da transcrição, por meio de uma estrutura secundária alternativa do transcrito proveniente da sequência líder, é chamada de atenuação e regula a expressão de operons envolvidos na biossíntese de aminoácidos. Esses operons codificam sequências líderes no RNAm produzido, que são traduzidas formando estruturas secundárias dependentes da tradução. No caso do Operon Trp, esse peptídeo líder de 14 aminoácidos possui dois códons para triptofano, no início da sequência, que exercem um papel muito significativo na regulação. Quando há pouco triptofano, o ribossomo fica parado nos códons UGG repetidos aguardando a chegada de um novo RNAt ligado ao triptofano. O ribossomo parado no início da molécula de RNAm (região denominada 1) permite que os nucleotídeos das regiões 2 e 3 façam estruturas em alças, não ocorrendo o término da transcrição pela falta de pareamento entre 3 e 4. Ao contrário, em altas concentrações de triptofano, as sequencias 3 e 4 fazem uma estrutura secundária em grampo (pareamento Gs e Cs) seguida de Us, indicando término da transcrição. A sequência líder do Operon Trp apresenta 162 nucleotídeos e está localizada antes do códon iniciador de trpE.
Vários outros operons para a biossíntese de aminoácidos em E. coli são hoje conhecidos como contendo sequências atuando como “atenuadores”. O peptídeo líder de cada um deles contém uma abundância de aminoácidos do tipo controlado pelo operon. Por exemplo, o operon de treonina codifica enzimas que sintetizam tanto treonina quanto isoleusina. O peptídeo líder contém oito treoninas e quatro isoleucinas em uma sequência de 16 aminoácidos. Sete fenilalaninas estão presentes no líder de 15 aminoácidos do operon da fenilalanina. Ainda mais marcantes são as sete histidinas em fila encontradas no peptídeo líder do operon de histidina.

