Unidade IV - Síntese de proteínas - Tradução
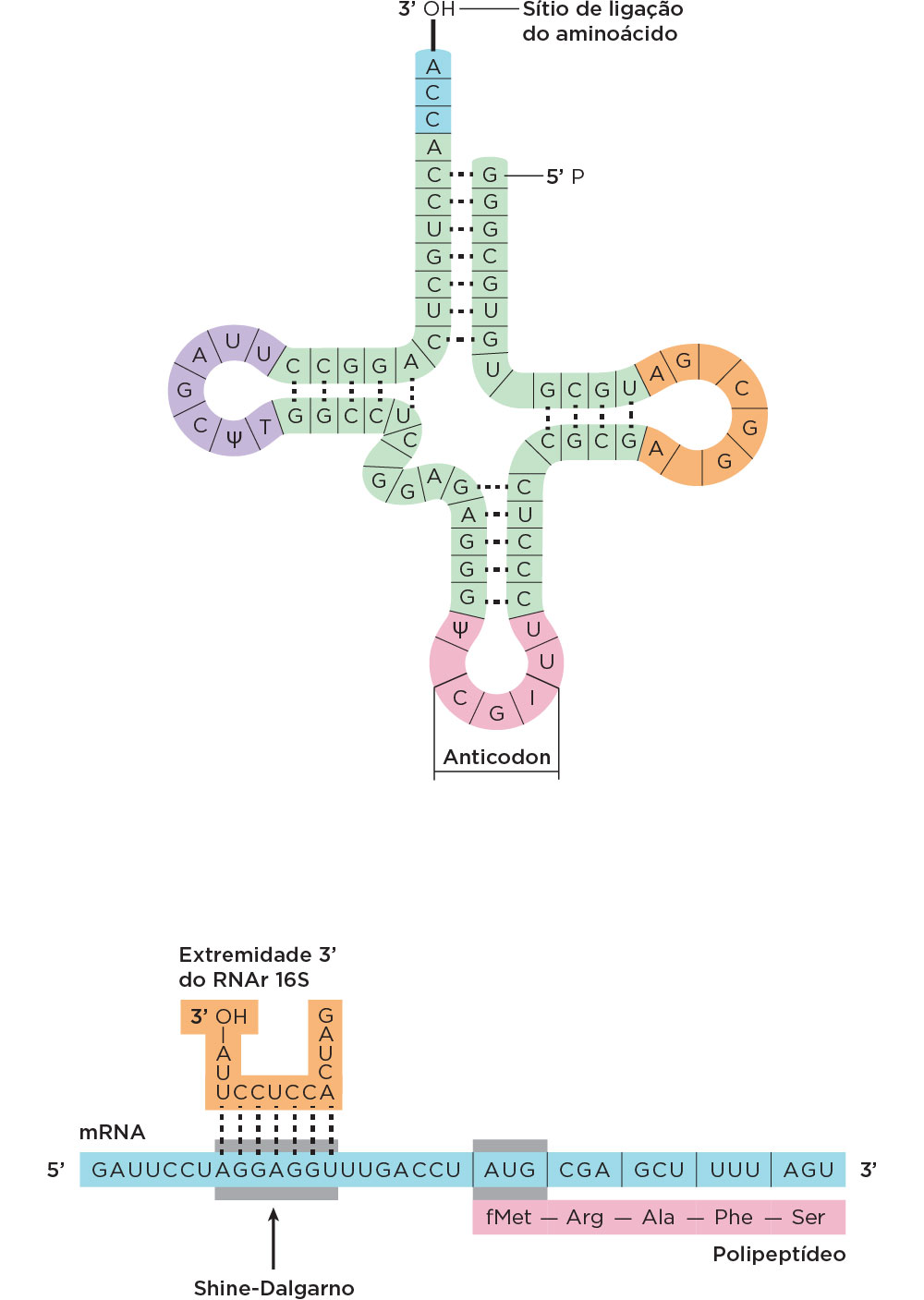
A síntese proteica envolve a ação dos três tipos de RNAs principais: o RNAm, o RNA transportador (RNAt) e o RNA ribossomal (RNAr). O RNAt possui uma estrutura tipo trevo de três folhas, com dobras em sua própria molécula formando em uma extremidade da dobre o anticódon, que corresponde à trinca de nucleotídeos que fará o pareamento com o códon do RNAm . Considerando o código genético que será descrito posteriormente nessa unidade, o códon dita o tipo de aminoácido a ser acoplado na região 3’ (término CCA) do RNAt, sendo que cada RNAt é específico para um determinado aminoácido. A interação entre RNAt (anticódon) e RNAm (códon) se dá por complementaridade de bases, sendo que a sequência de aminoácidos para um polipeptídeo (ou proteína) é dada pelo códon presente no RNAm.
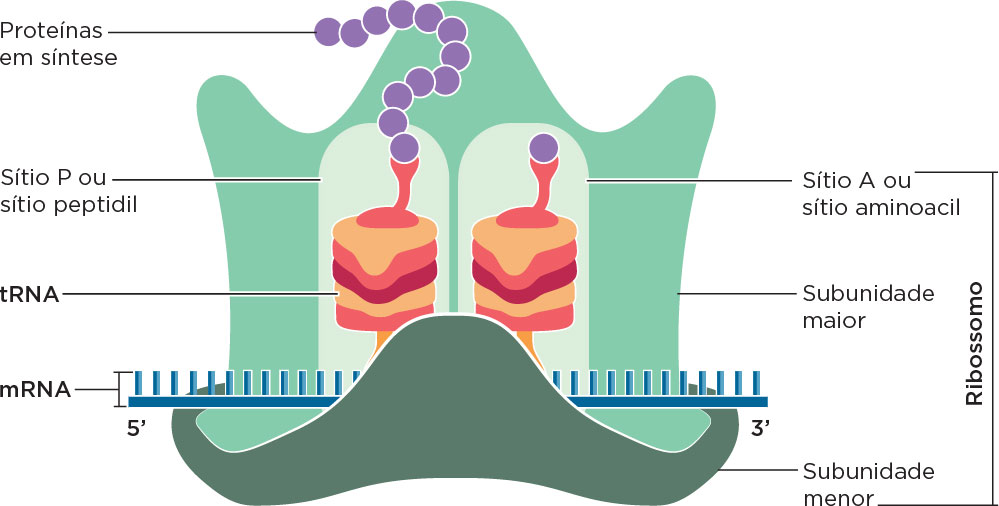
Diferentes tipos de RNAr compõem os ribossomos, a organela responsável pela síntese proteica. Em eucariotos, os RNAr de coeficiente de sedimentação 28 S e 18 S são os principais na composição do ribossomos associados a outros RNAr menores e proteínas formam as subunidades ribomossomais. Os principais em procariotos são do 26 S e 13 S. No início da formação do complexo para a síntese proteica ocorre a interação da subunidade ribossomal menor com o RNAm e, na subunidade maior, formam-se o peptídeo pela carregamento de aminoácidos pelo RNAt, que ocupam os sítios aminoacil (A) seguido do peptidil (P) presentes nessa subunidade. Em procariotos, à medida que o RNAm vai sendo transcrito, também vai sendo traduzido. Já em eucariotos isso não ocorre, pois o RNAm deve migrar do núcleo para o citoplasma para que seja traduzido, ou seja, não são eventos acoplados.
No RNAm existem sequências que sinalizam para início e término de tradução. O códon iniciador de tradução é o AUG, que codifica para uma metionina em eucariotos e para uma formilmetionina em procariotos. Em procariotos, o RNAm apresenta uma sequência localizada a cerca de sete nucleotídeos acima do códon AUG que se pareia com sequências consenso do RNAr 16 S, chamada de Shine-Dalgarno (ou RBS, ribosome binding site). O primeiro RNAt liga-se à subunidade menor e o conjunto desliza pelo RNAm até encontrar o códon AUG. Para cada RNAm existem potencialmente três diferentes leituras codificantes (já que as bases são lidas em trincas), porém só uma das combinações de leitura (ORF, do inglês open reading frame) é correta sendo que o provável produto proteico frequentemente é a cadeia de polipeptídeos mais longa. Assim, a primeira trinca AUG abaixo da sequência de Shine-Dalgarno será o início da síntese proteica .
Ao conjunto subunidade ribossomal menor-RNAt na posição exata para início da síntese proteica liga-se a subunidade maior, completando o ribossomo. O sítio A do ribossomo vazio aguarda a chegada do próximo RNAt, transportando o aminoácido correspondente ao códon apresentado após o códon AUG. Quando o RNAt traz esse novo aminoácido e se liga ao RNAm acontece a reação química chamada de ligação peptídica entre o primeiro aminoácido e o segundo. O primeiro RNAt que ocupava o sítio P do ribossomo se desliga, podendo transportar um novo aminoácido. O ribossomo então desloca-se no mesmo sentido 5’→3’ do RNAm ocupando um novo codón o que libera o sítio A para ser ocupado por novo RNAt trazendo o aminoácido de acordo com a sequência do códon .
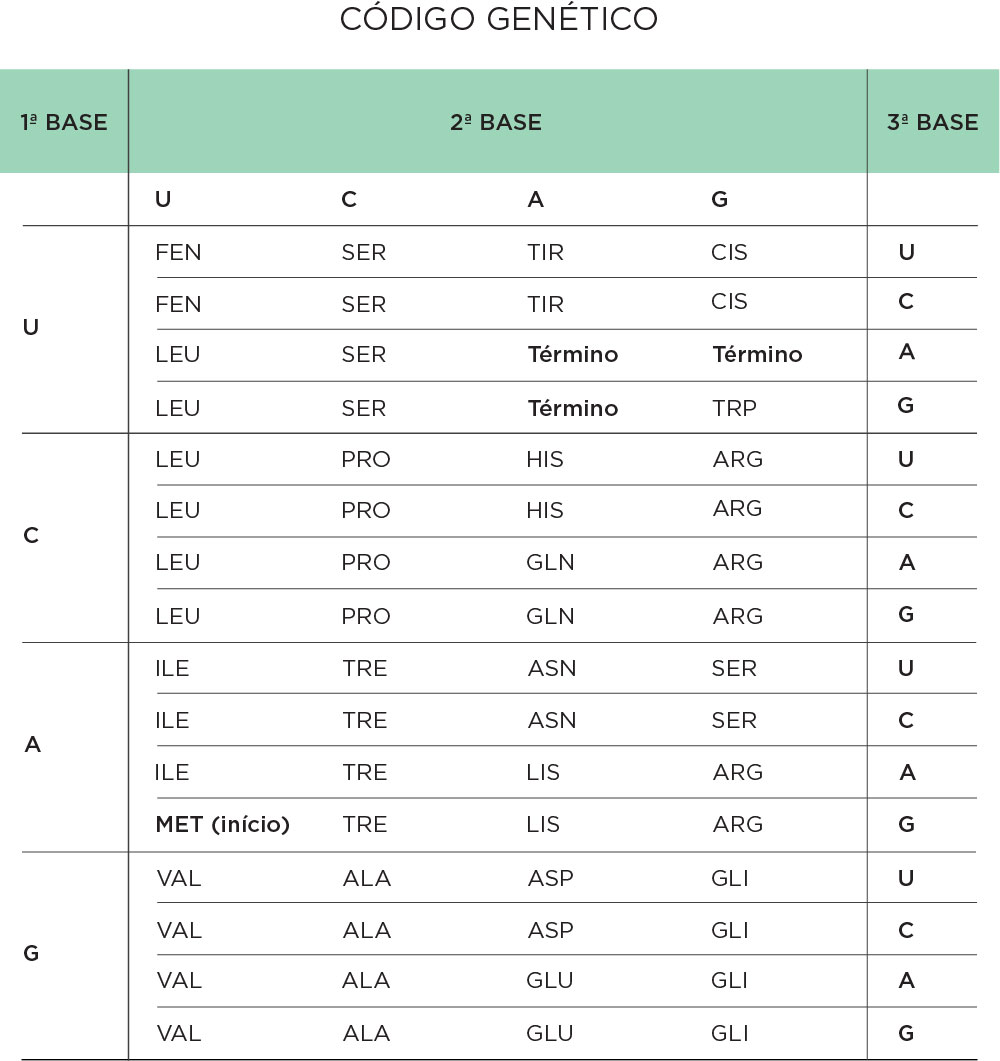
O código genético responsável por elucidar qual aminoácido deve ser trazido pelo RNAt de acordo com a sequência codificante presente no RNAm foi completamente elucidade em 1966, pelos pesquisadores Marshall Nirenberg e Heinrich Matthaei, trabalhando com triplets de UUU, com a maquinaria de tradução de E.coli e com aminoácidos radioativos observaram que poli Us codificava para o aminoácido fenilalanina. Os experimentos posteriores, envolvendo diferentes combinações de bases nos códons, permitiram decifrar o código genético. Dentre as características apresentadas pelo código genético estão o fato de ser degenerado ou redundante, ou seja, um mesmo aminoácido pode ser codificado por códons diferentes; é universal, com poucas exceções é o mesmo nos mais diversos organismos e não é superposto, por ser lido em trincas sequencialmente .
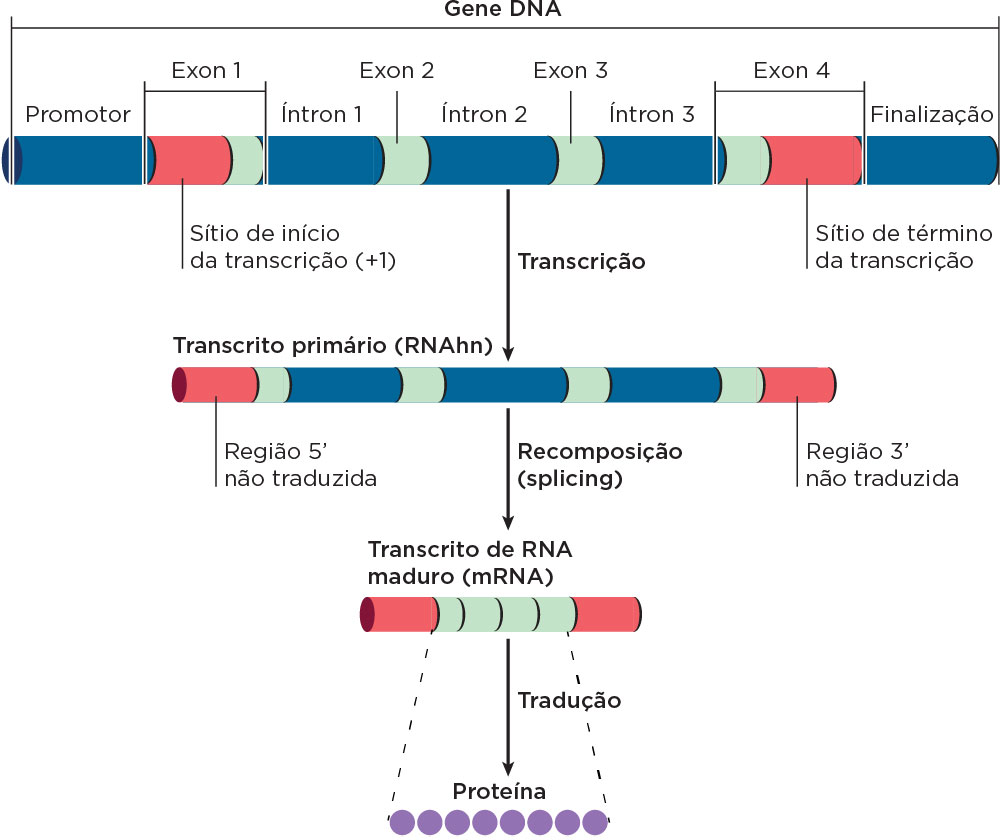
O processo de movimento do ribossomo pela fita de RNAm se repete até que um dos três (UAA, UAG, UGA) sítios de terminação (códons de término ou stop codons) seja encontrado nessa fita. Neste caso, o ribossomo espera não um RNAt mas uma proteína conhecida como fator de liberação (RF, do inglês releasing factor), que se liga no sítio A ao códon de terminação, desestabiliza o ribossomo desfazendo a maquinaria traducional. Há vários fatores proteicos chamados fatores de iniciação e de alongamento que também colaboram no processo de síntese proteica. O intervalo de sequência do RNAm, localizado entre a posição +1 do RNAm transcrito e o códon AUG de início de tradução, é chamado de região 5’-UTR e a porção que não será traduzida, localizada após o stop códon, é chamada de 3’-UTR. Ambas as sequências não traduzidas podem estar presentes em transcritos de genes de procariotos e eucariotos. Considerando a sequência codificante no RNAm descrita na Figura 33, observe as possibilidades de leitura. A provável ORF é aquela mais longa começando por um AUG até um stop códon. A Figura 34 apresenta uma visão geral do fluxo da informação genética, considerando as sequências sinais específicas.