Unidade III - Síntese de RNA - Transcrição
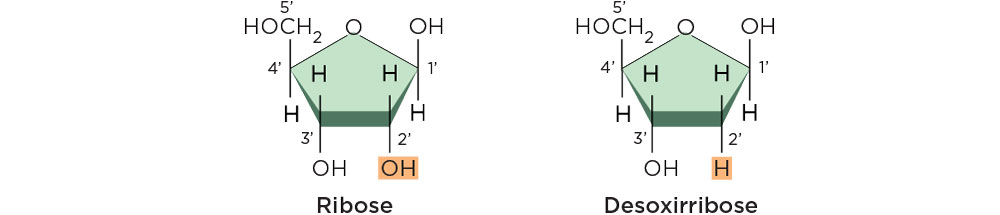
A transcrição consiste na síntese de uma molécula de RNA a partir das informações contidas na sequência de nucleotídeos de uma molécula de DNA. A transcrição é um processo extremamente variável, sendo a célula quem controla quando determinadas sequências devem ser transcritas e o quanto de RNA deve ser sintetizado. Esse mecanismo é conhecido como Controle ou Regulação da Expressão Gênica. Assim como no processo de replicação, a transcrição ocorre no sentido 5’ ® 3’ e é baseada na complementaridade de bases, sendo que na formação do RNA apenas uma fita de DNA é usada como molde, o açúcar da pentose é uma ribose (hidroxila no carbono 2 da pentose), o nucleotídeo uracila (U) substitui a timina no pareamento com a base adenina e o RNA formado é unifilamentar (fita simples), com a mesma sequência da fita não-molde do DNA, também chamada de codificante, trocando o nucleotídeo T por U no RNA .
Dentre os aspectos diferenciais entre replicação e transcrição, destacam-se que esse último processo não requer um primer iniciador da reação de polimerização, apenas uma das fitas de DNA é copiada e a enzima responsável pela transcrição é a RNA Polimerase, que tem por função desnaturar as fitas de DNA, manter as fitas separadas, estabilizar o híbrido DNA/RNA, renaturar as fitas de DNA e terminar a síntese de RNA.
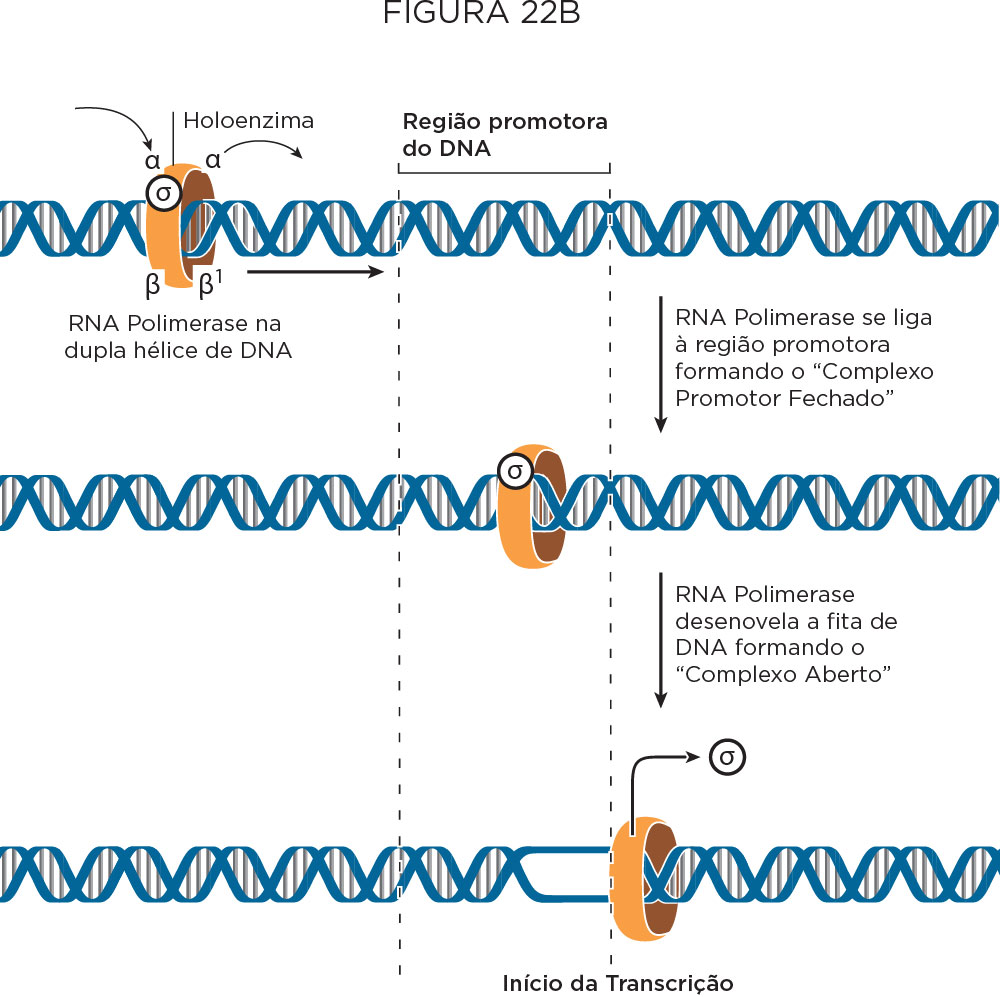
O processo de transcrição envolve três fases. Na fase de iniciação em bactérias, a RNA Polimerase inicia a cadeia nova por meio da formação de uma “bolha” de separação das fitas de DNA de forma dependente de sequências promotoras. Os promotores são sequências específicas de reconhecimento para a RNA Polimerase, que sinalizam exatamente onde a síntese de RNA deve ser iniciada, sendo altamente conservadas entre diferentes genes e espécies. O promotor antecede o sítio de iniciação de transcrição, que por convenção, a primeira base a ser transcrita é numerada como +1 .
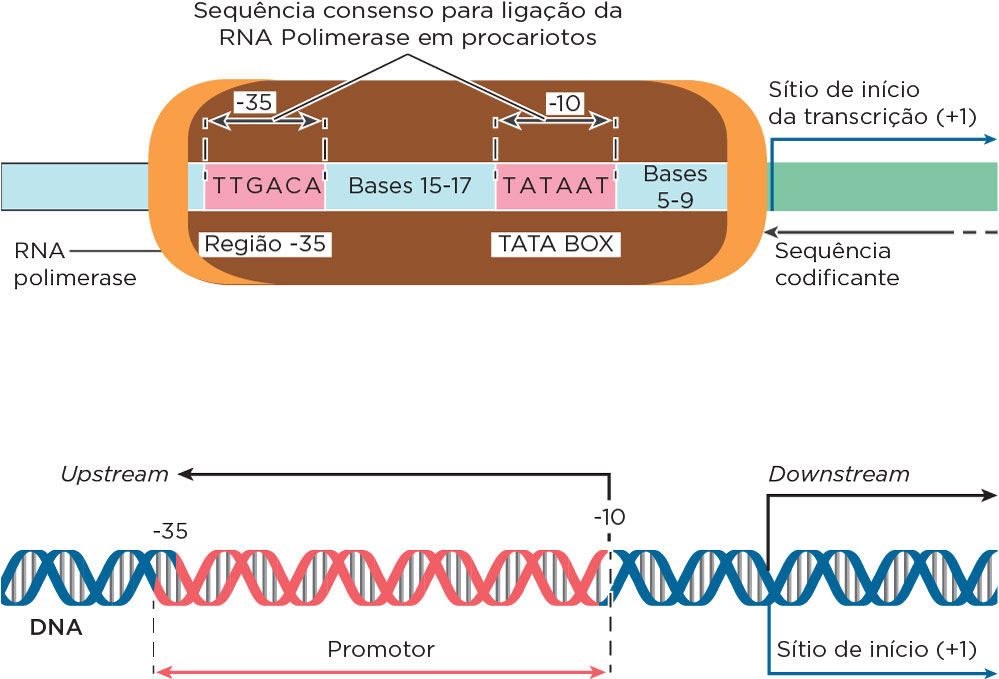
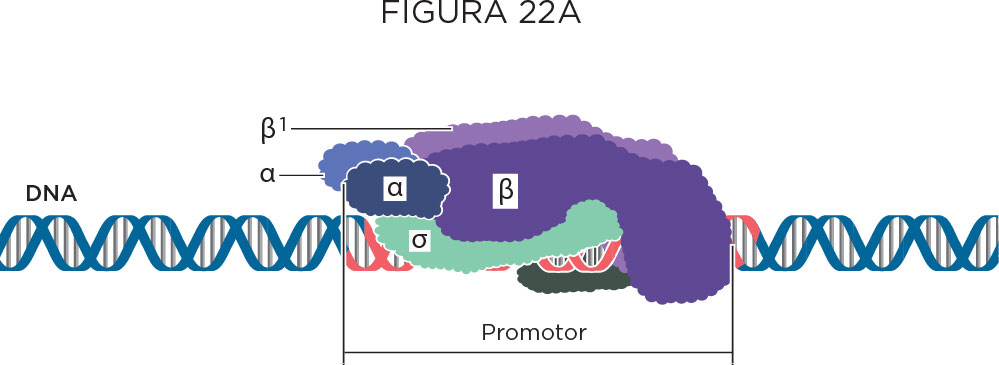
Nessa fase de iniciação, a RNA Polimerase, uma holoenzima composta por cinco subunidades, faz contato com as regiões promotoras e deselicoidiza o DNA pela ação de sua subunidade dissociativa, o fator sigma. O fator sigma reconhece a região promotora, formando o Complexo Promotor Fechado. Após promover a deselicoidização de bases próximas à região -10 da fita de DNA que contém a região promotora, dá-se a formação do Complexo Promotor Aberto pela liberação do fator sigma e início da transcrição .
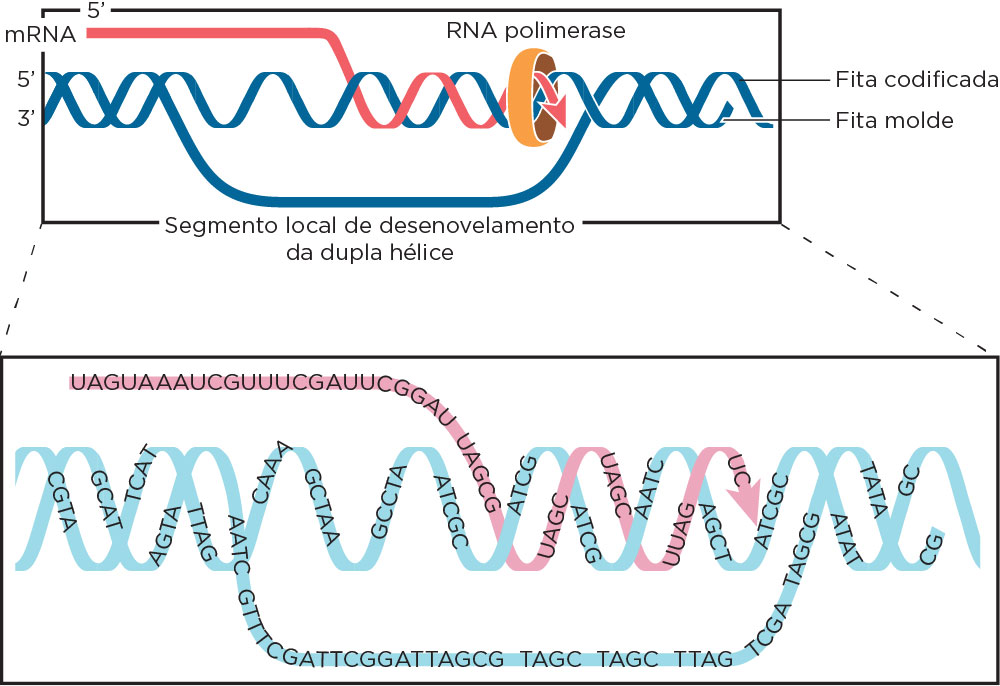
Após dissociação do fator sigma se inicia a segunda fase do processo de transcrição, o alongamento (ou elongamento), que é baseado na incorporação de nucleotíedeos pela RNA polimerase no sentido 5’ ® 3’ por complementaridade de bases. A “bolha de transcrição” é mantida e se move ao longo do duplex de DNA enquanto a RNA Polimerase vai adicionando os ribonucleotídeos e “construíndo” a fita simples de RNA .
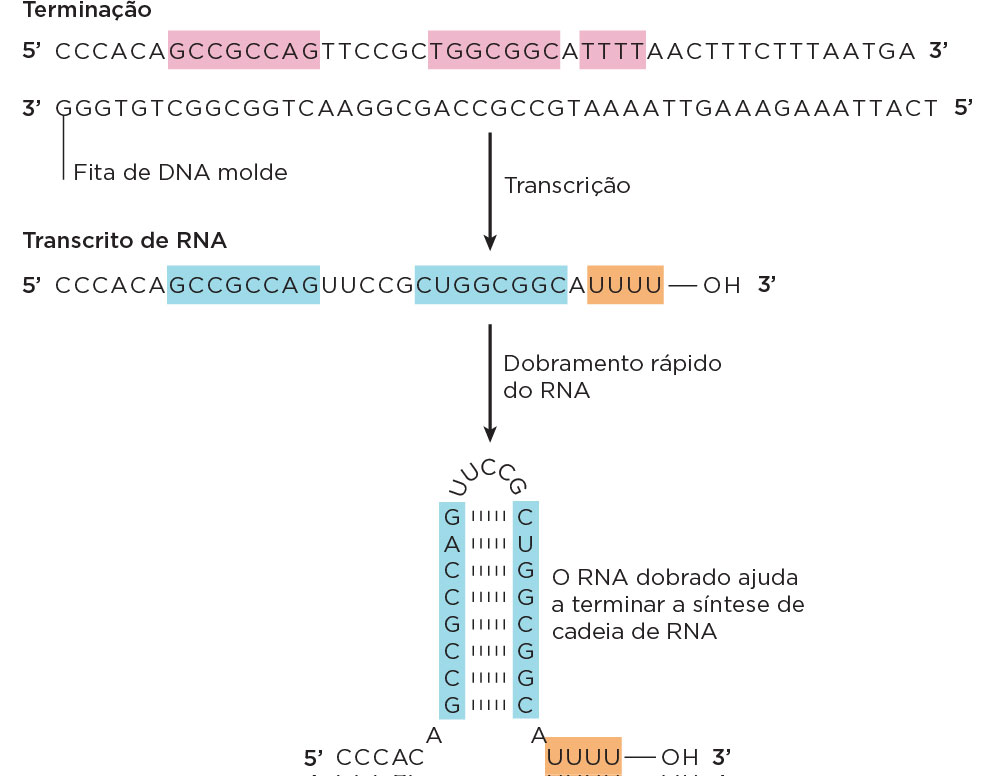
O término da transcrição instrínseco se dá pela formação de motivos estruturais envolvendo cerca de 40 bases terminais, baseados em sequências de bases repetidas e invertidas ricas em Cs e Gs e à formação no RNA nascente de uma alça ou grampo de terminação, seguido de uma sequência rica em U. Essa sequência de poli Us pode facilitar o desligamento da RNA Polimerase do complexo transcricional devido à fraca ligação entre A e U.
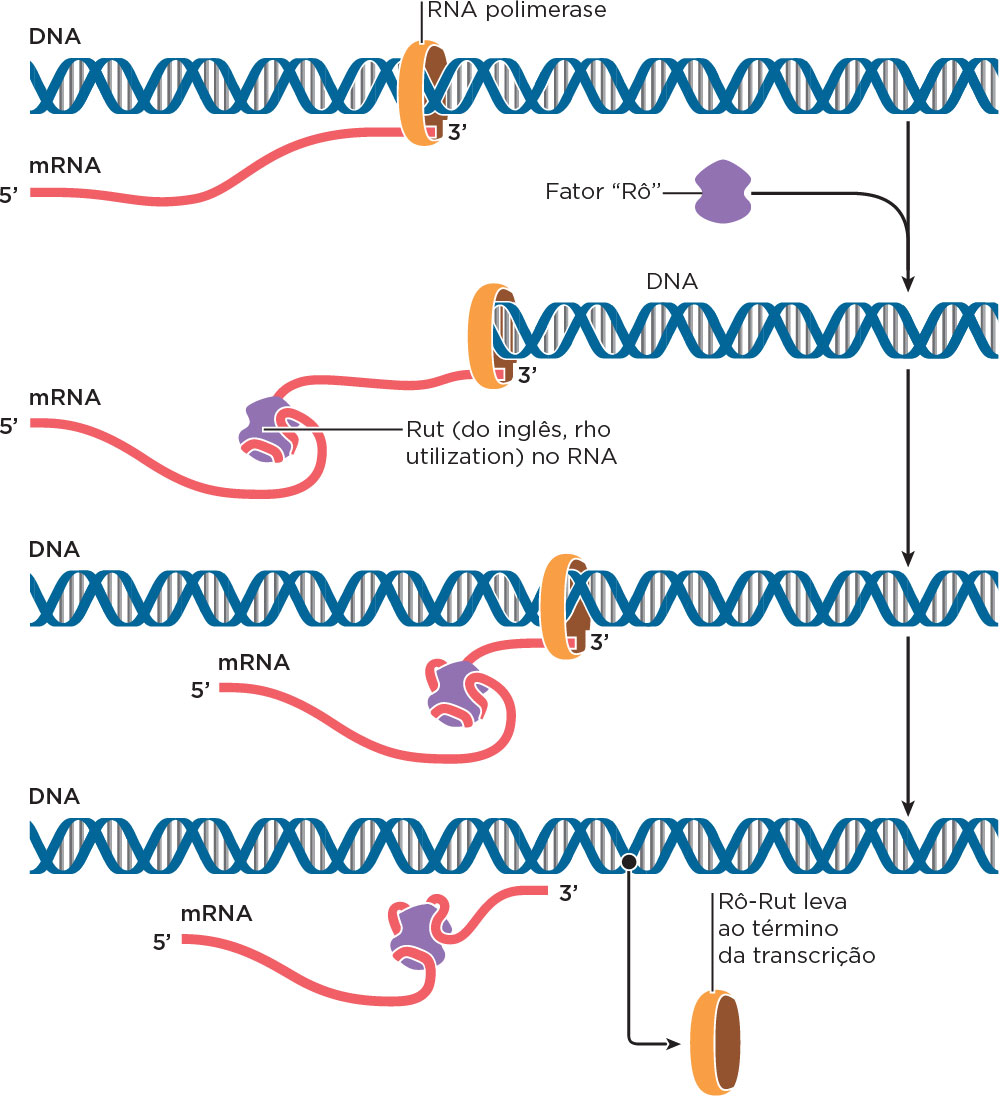
Para RNAs que não apresentam esses motivos, existe um segundo tipo de mecanismo de término, conhecido como Rô-Rut. A proteína denominada Rô reconhece sinais de término, os quais geralmente não têm sequências capazes de formar grampo na extremidade 3’ do RNA nascente, sendo que esses RNAs podem conter uma sequência rica em C mas pobre em G, com segmento posterior chamado denominado Rut (do inglês, rho utilization). A proteína Rô, dependente de ATP, move-se mais rapidamente sobre a fita de RNA nascente do que a RNA Polimerase, e ligando a sequência Rut leva à dissociação do RNA do complexo de transcrição (.
Considerando que os genomas eucarióticos são maiores e com mais genes a serem reconhecidos e transcritos, existe uma divisão do trabalho de transcrição entre três RNA polimerases, sendo que de forma simplificada, a RNA Polimerase I transcreve RNA ribossomal (RNAr), a do tipo II transcreve RNA mensageiro (RNAm) e alguns snRNAs (pequenos RNAs nucleares) e a do tipo III transcreve RNA transportador (RNAt) e alguns snRNAs. Os promotores eucarióticos não são capazes de fornecer sinais de reconhecimento suficientes para que a RNA Polimerase inicie a transcrição. Os promotores eucarióticos são inicialmente reconhecidos por proteínas regulatórias chamadas de Fatores Gerais de Transcrição (GTFs ou simplesmente TFs – do inglês, transcription factor).
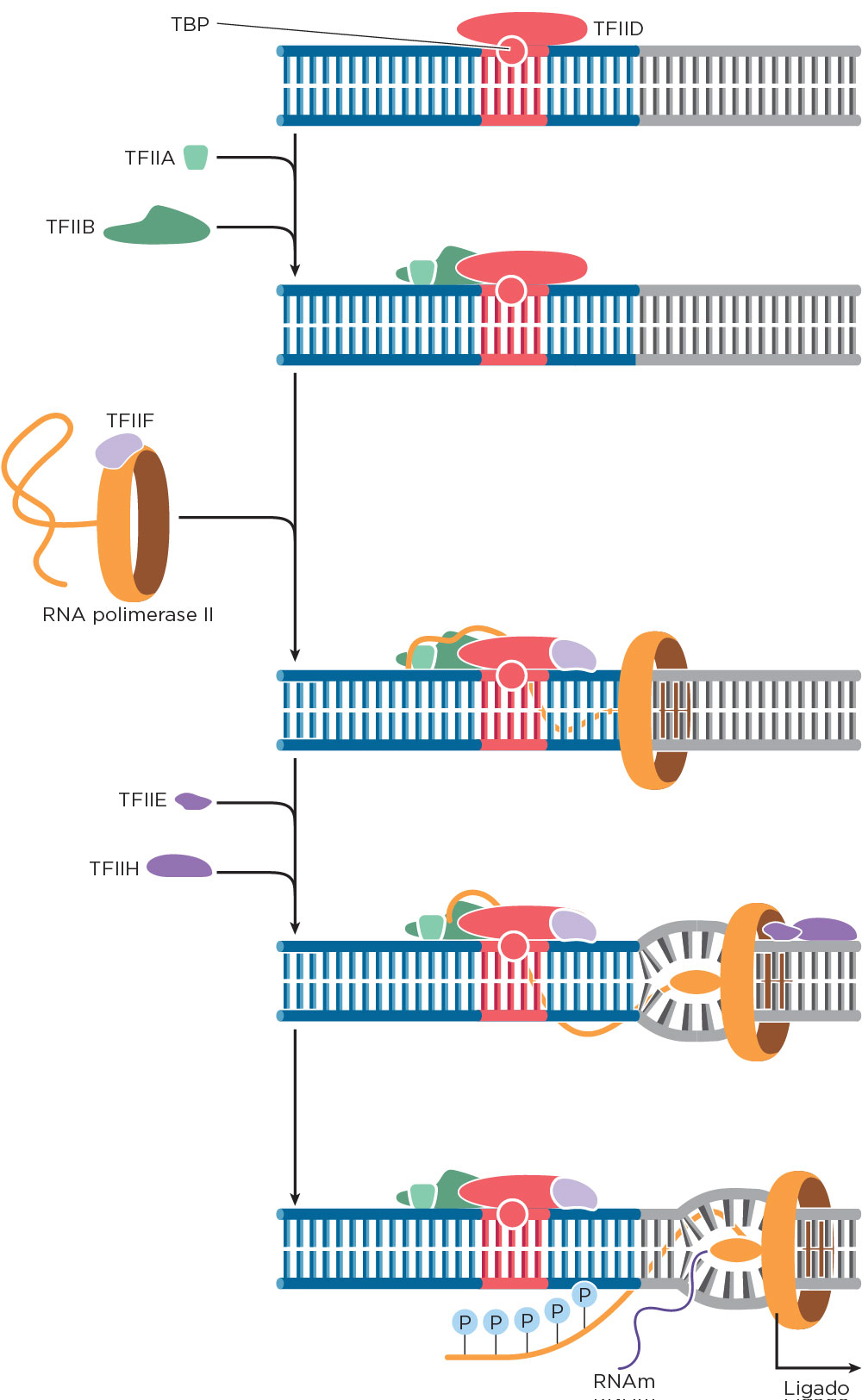
Na fase de iniciação de transcrição os TFs reconhecem as sequências promotoras eucarióticas, em que o motivo TATA boxe quando presente no gene ocupa a posição relativa de -25 pb do sítio +1, além de existir outras sequênciais proximais CCAAT, Gs/Cs ou motivos em octâmeros ocupando variadas posições relativas a partir do sítio +1. Para transcrição de RNAm, os TFs recebem a denominação de TFII em função do tipo de RNA Polimerase responsável pela síntese do RNAm. Os TFIIs que interagem com a RNA Polimerse II é representado por seis proteínas principais, sendo todas essas um complexo multiprotéico.
Em ordem, o primeiro evento na formação do complexo de pré-iniciação de transcrição consiste na ligação da proteína de ligação ao TATA (TBP, do inglês TATA binding protein), uma subunidade do complexo TFIID. Após essa ligação ao TATA, os outros TFIIs (TFIIA, TFIIB, TFIIF, TFIIE e TFIIH) e a polimerase são recrutados, juntamente com outras proteínas, se ligando de forma coordenada para haver desnaturação do DNA, por meio da ação da TFIIH e hidrólise de ATP (semelhante à ação da DNA helicase). Para passar para a segunda fase do processo de transcrição, o alogamento, a RNA Polimerase II é fosforilada no seu domínio carboxi terminal (CTD) que apresenta sequência repetida composta por aminoácidos, tais como, a serina e a prolina, os quais apresentam sítios de fosforilação por quinases específicas, função que pode ser atribuída a uma das subunidades do TFIIH que é uma quinase. Assim, o TFIIH é o mais complexo comparado aos demais fatores, apresentando subunidade que atuam como ATPases e outra como proteinaquinase. O estado fosforilado no CTD da RNA Polimerase II permite a liberação dessa enzima do complexo de proteínas de pré-iniciação, a qual deixa o promotor e inicia o alongamento .
A fase de alongamento ocorre na bolha de transcrição e diferente do que ocorre com procariotos, em que à medida que o RNA é transcrito vai sendo traduzido; em eucariotos, alguns eventos terão que ocorrer com o RNAm no núcleo celular, antes da tradução no citoplasma. As etapas associadas à maturação do RNAm formado é conhecida como Processamento do RNA.
As etapas do processamento do RNA heterogêneo nuclear (RNAhn) precursor do RNAm envolvem a modificação do RNAm nascente na extremidade 5’ pela adição de um capacete de 7-metil-guanosina, que tem por função a proteção desse RNA contra o ataque de nucleases e para o direcionamento dos ribossomos sobre essa extremidade do RNAm quando está no citoplasma. Na região intermediária do RNAm também ocorre alterações importantes na função que essa molécula irá exercer na célula. Essa modificação envolve um processo chamado de splicing, que envolve a retirada de regiões não codificantes ou intercalares chamadas de introns e junção das sequências codificantes, os exons. A última modificação ocorre na extremidade final do RNAm, a região 3’, na qual ocorre a adição de uma cauda poli A (cerca de 200 As) pela ação da enzima Poli A Polimerase .
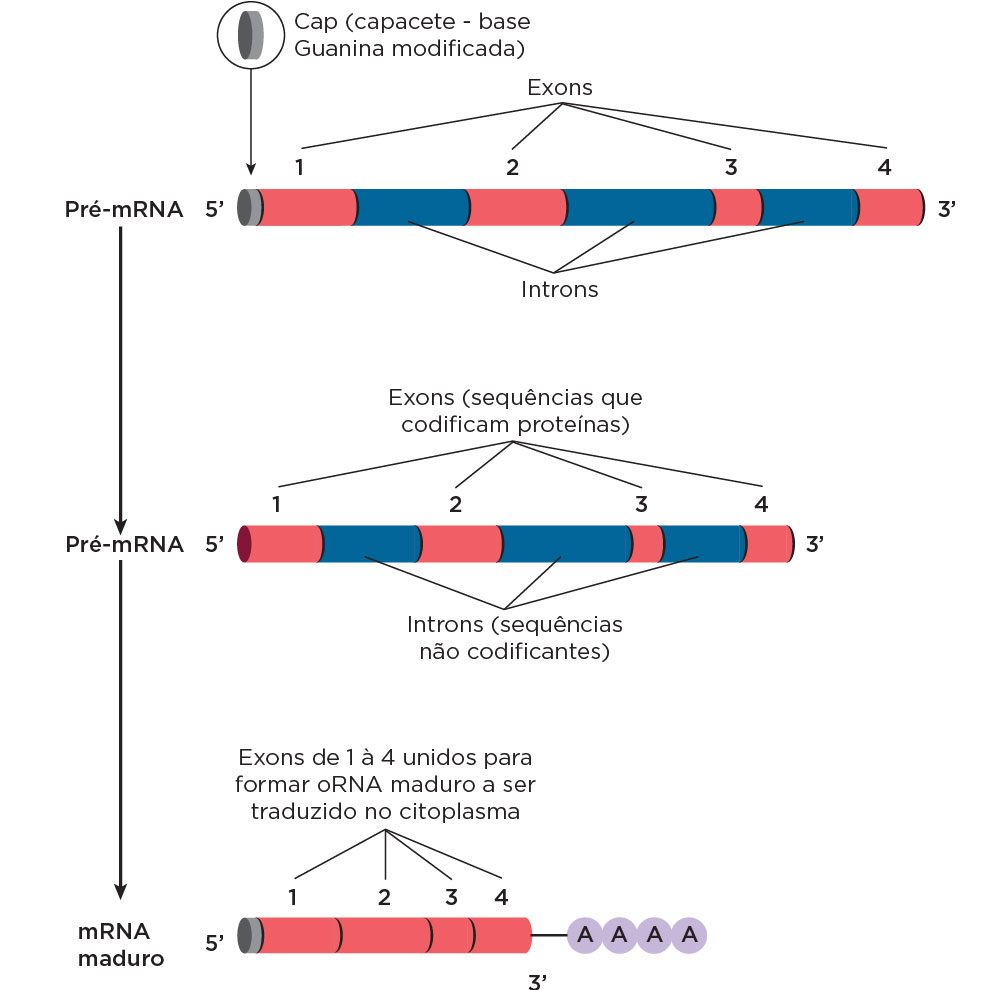
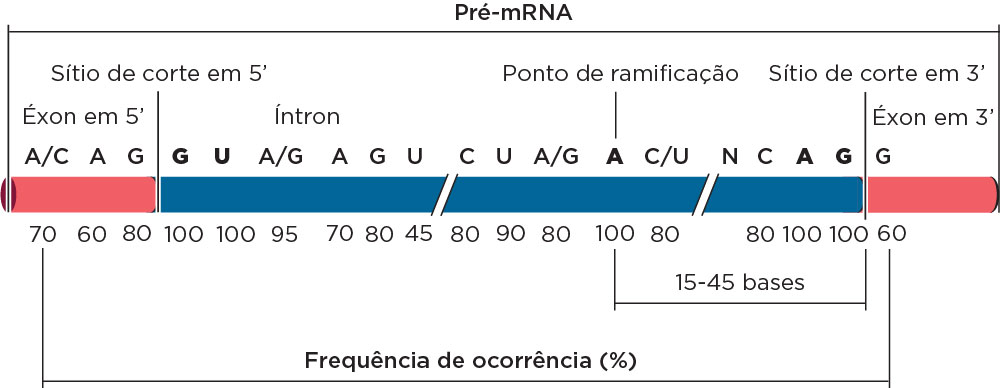
O splicing é realizado por um complexo formado por pequenos RNAs nucleares (snRNAs), associados a proteínas (snRNPs), chamado de spliceossomo. O spliceossomo ajuda a clivar o RNAm nos sítios doadores e aceptores de splicing, remove os introns, impede o afastamento dos exons, e, por fim, une os exons. A junção por meio do splicing requer a presença de sequências conservadas de bases AG no término do exon, GU no início e AG no término do intron, iniciando o próximo exon com GU. Nessas junções alguns nucleotídeos específicos foram encontrados sendo quase idênticos entre os genes e entre as espécies. O sítio A, chamado de ponto de ramificação, situa-se a cerca de 15-45 nucleotídeos antecedentes à 3’ do ponto de corte no intron . A retirada do intron se dá pelo reconhecimento das regiões terminais dos exons e dos introns, formando uma dobra em sua própria molécula (alça), com participação do ponto de ramificação e das snRNPs que se associam ao mRNA propiciando o corte e novamente a junção.
A etapa de processamento é dita co-transcricional, pois ocorre nas três regiões do RNA à medida que a síntese vai acontecendo. Além do processamento que se dá por spliceossomo, existem moléculas de RNAs capazes de fazer a sua própria recomposição, chamadas de introns de auto-recomposição (self-splicing). Estudos recentes demonstram que os snRNAs e não o componente protéico do spliceossomo são os responsáveis pela reação de catálise para remoção dos íntrons. Os RNAs com essa capacidade catalítica, tais como, os RNAs do spliceossomo e várias outras moléculas de RNAs são chamados de ribozimas, pois apresentam muitas das características de uma enzima clássica como sítio ativo, sítio de ligação para substrato e sítio de ligação para um cofator (como um íon metálico).
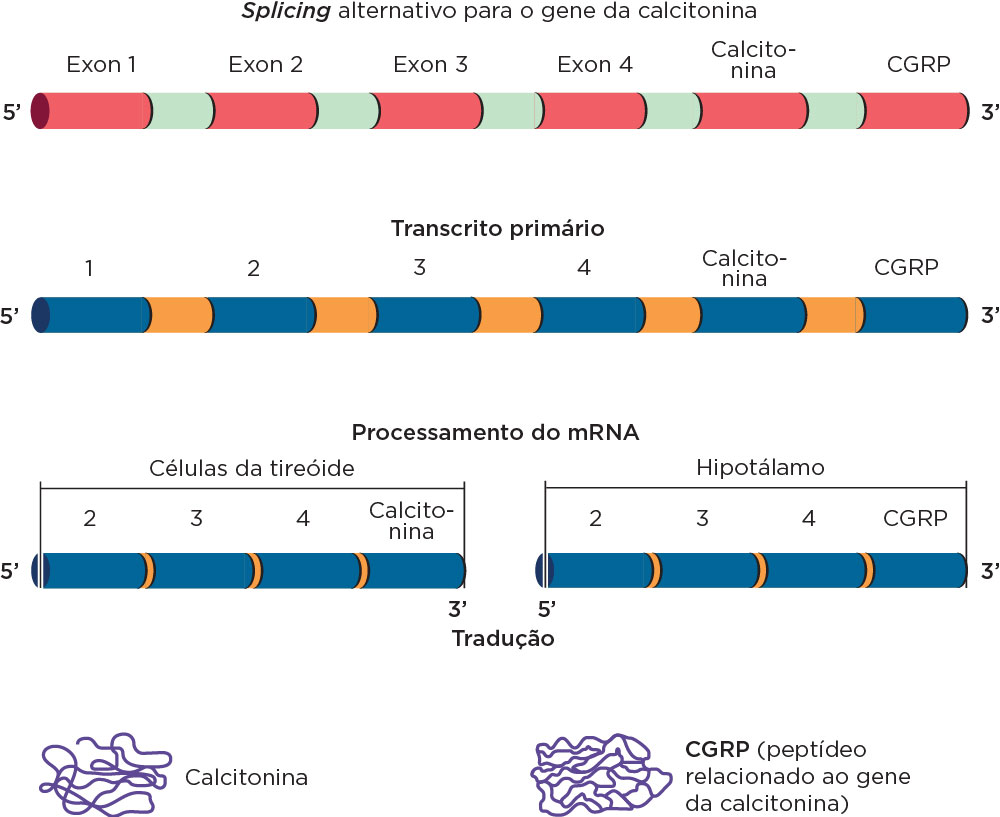
No que se refere ao término da transcrição nos eucariotos em muitos genes pela presença de um sinal composto pelas bases AAUAAA ou AAUUAAA (sinal de poliadenilação) na extremidade 3’ do RNAm. Como será observado na unidade seguinte, após sintetizado, o RNAm não apresenta qualquer associação com a tradução, ou seja, o tamanho do RNAm não corresponde ao tamanho da proteína a ser traduzida, pois além do splicing, o RNAm apresenta sequências transcritas, mas não traduzidas tanto na extremidade 3’ como na 5’ (UTRs, do inglês untranslated region). Outras características apresentadas por alguns tipos de RNAs, é a possibilidade de sofrer splicing alternativo, que se refere à recomposição sequencial alternativa dos exons e ocorre para atender uma necessidade tecido-específica.
Um exemplo de splicing alternativo pode ser observado para o gene da calcitonina. O RNAm recomposto para a expressão da proteína calcitonina, que atua nas células da tireoide, apresenta exons diferentes da calcitonina (CGRP – calcitonin gene related peptide) expressa pelas células do hipotálamo .