Unidade II - Replicação do DNA
A estrutura do DNA é simples quando comparado à informação por ele carregada necessária para a manutenção de todas as atividades celulares. Como vimos na unidade anterior, a molécula de DNA é um polímero formado por monômeros chamados nucleotídeos (ou mais especificamente desoxirribonucleotídeos). A fita de DNA é dupla, com exceção de certos vírus. As duas fitas são antiparalelas entre si e as bases de cada fita são complementares. As interações entre bases acontecem pela interação do tipo ligações de hidrogênio (“pontes”), sendo que a base A (adenina) interage com T (timina) se for DNA-DNA, ou com U (uracila) se for DNA-RNA por duas pontes de hidrogênio; a G (guanina) interage com a C (citosina) por três pontes de hidrogênio.
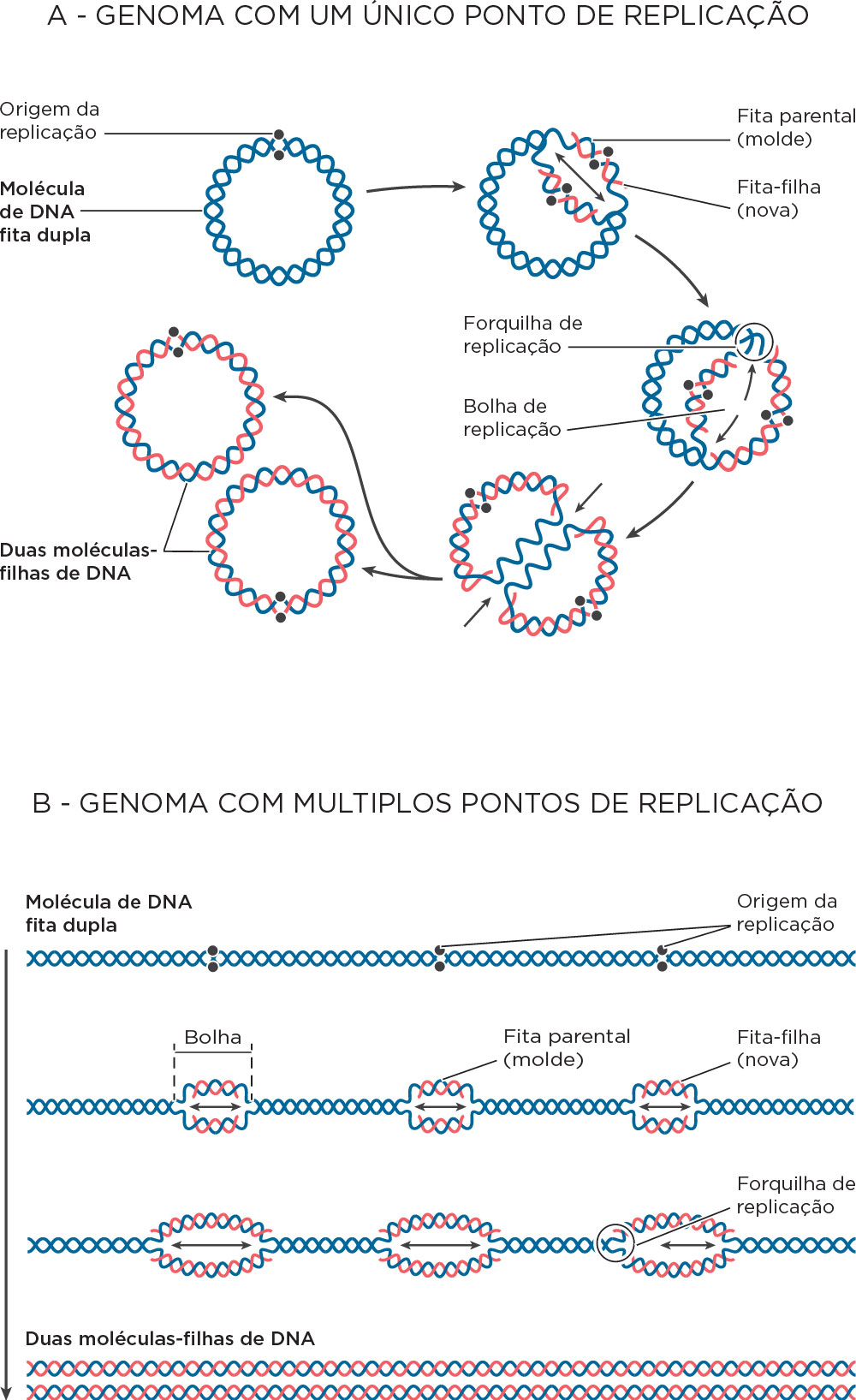
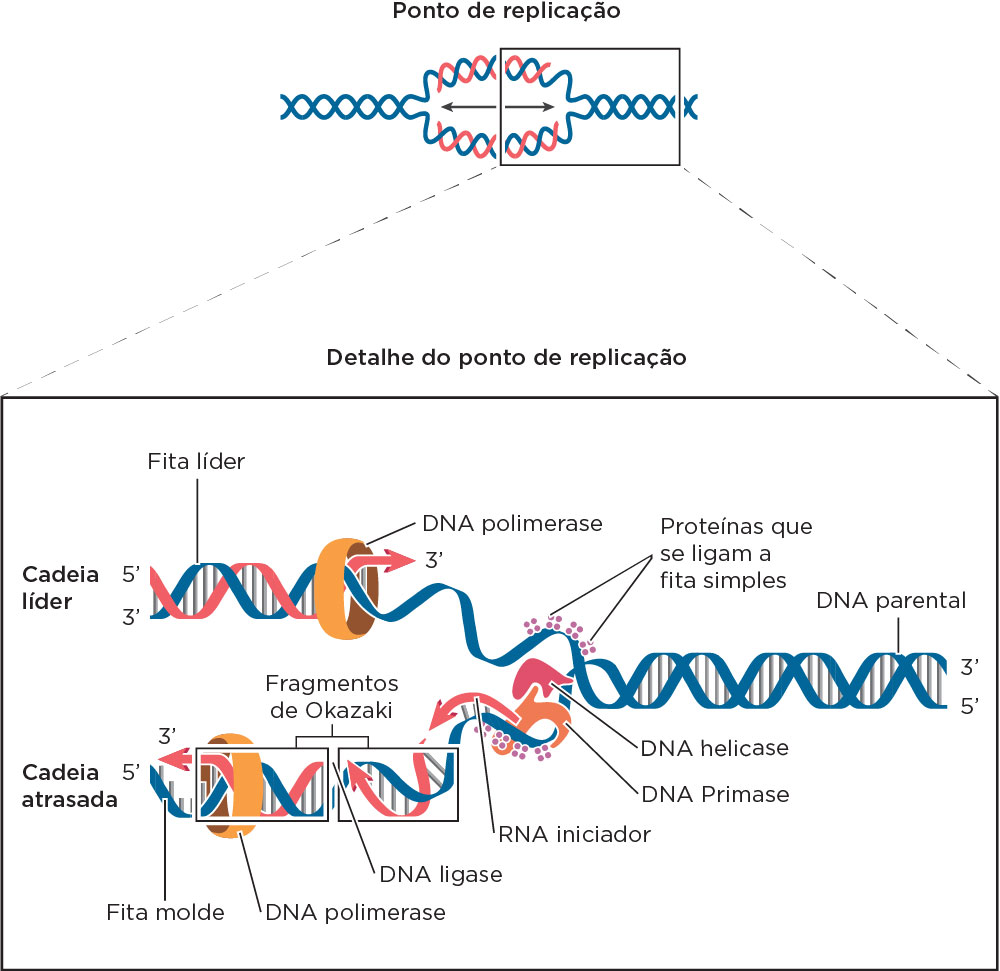
Na replicação do DNA, ambas as fitas, paralela e antiparalela, devem ser replicadas ao mesmo tempo e, portanto, em direções opostas. Dessa forma, a partir do processo de replicação, que ocorre birecionalmente, são formadas duas fitas novas, uma contínua (leader) e uma descontínua (lagging), em função da sua visualização a partir de um ponto de simetria. Ou seja, a partir desse ponto de simetria ocorre a inversão entre as fitas, sendo que a líder para ser descontínua, e vice-versa. Cada fita alvo (template) age como molde para a síntese da nova fita, sendo a replicação do tipo semiconservativa. O início do processo se dá pela ação da DNA helicase, a qual desenovela o DNA na origem ou forquilha de replicação utilizando energia do ATP, transformando o DNA de dupla fita em DNA de fita simples (single strand DNA – ssDNA) e permitindo que cada uma das fitas simples seja copiada.
Assim, a replicação requer atividade coordenada de diversos tipos de enzimas, dentre estas, estão as com atividades de topoisomerases (promovem quebras temporárias para remover o superenovelamento do DNA no local da replicação, deixando-o mais relaxado), helicases (levam à deselicoidização da dupla hélice na forquilha de replicação e rompimento das ligações de hidrogênio, que unem os dois filamentos de DNA na dupla hélice), primases (adicionam os “primers” ou iniciadores - oligonucleotídeos de RNA - fornecendo a porção 3’-OH livre para a incorporação de novos nucleotídeos por complementaridade de bases pela DNA Polimerase), DNA polimerases (do tipo III adiciona nucleotídeos aos primers preexistentes para a formação das novas fitas e adiciona os nucleotídeos na fita retardatária após a retirada dos primers pela DNA Polimerase do tipo I), proteínas SSB (single strand binding protein – proteínas de ligação ao DNA de fita simples, que tem função de estabilizar o DNA deselicoidizado se ligando ao DNA unifilamentar e retardando a reconstituição de dúplex), DNA ligases (fazem a ligação fosfodiéster entre os fragmentos de Okazaki da fita descontínua para formar uma fita contínua, após remoção dos primers e adição de nucleotídeos pela DNA Polimerase III).
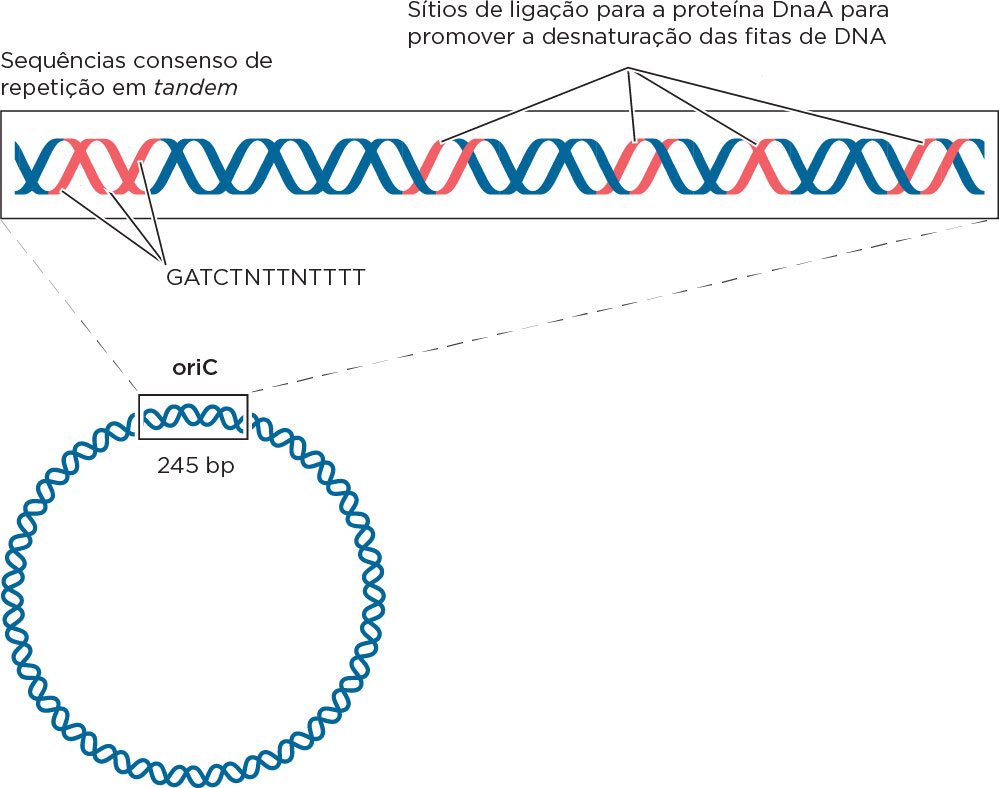
Como dito anteriormente, no cromossomo de procarioto o processo se inicia a partir de uma origem de replicação chamada de ori C, que apresenta um tamanho aproximado de 245-pb cuja sequência é rica em bases A e T ; enquanto em eucariotos existem várias origens, por vezes citadas como ARS (Autonomolously Replicating Sequence - sequências de replicação autônoma). Essas sequências têm sido mais bem compreendidas em leveduras (Saccharomyces cerevisae), mas é assumido que existe em todos os cromossomos eucarióticos. A replicação pode se iniciar simultaneamente em nos ARS dispostos ao longo dos cromossomos durante o processo de replicação, formando um processo cooperativo e rápido de replicação bidirecional .
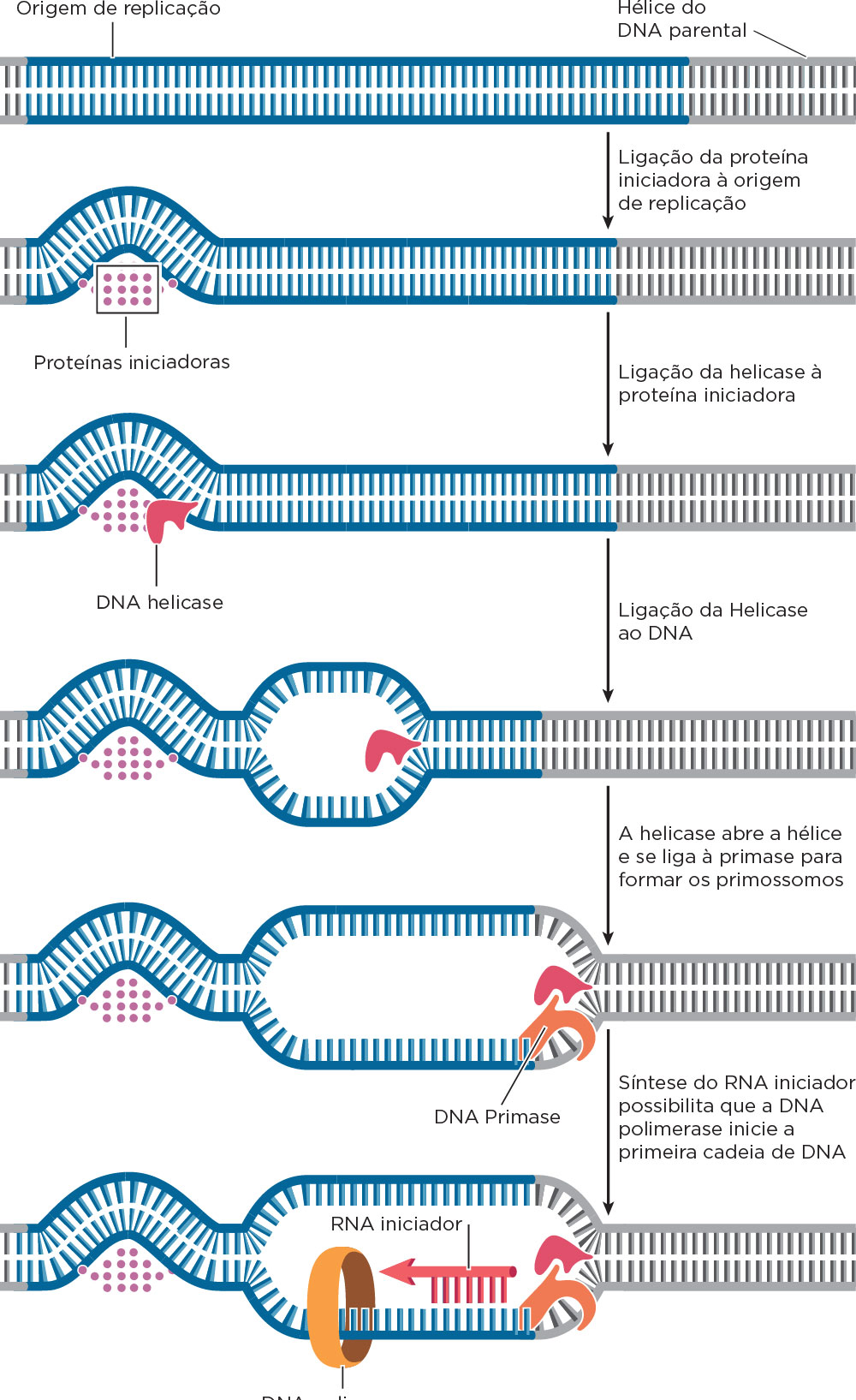
Em síntese, uma vez abertas as fitas do DNA, é necessário que essas permaneçam abertas para que a replicação ocorra. As proteínas responsáveis pela prevenção do reanelamento das fitas de DNA são chamadas de SSB mantendo o DNA em fita simples durante o processo de replicação. A primase sintetiza um primer de RNA, e, juntamente, com a DNA helicase e outros polipeptídeos forma o primossomo . O primer servirá como material inicial para a adição de nucleotídeos pela DNA Polimerase III, fornecendo uma extremidade com 3’-OH livre. Por ser um processo bidirecional, depois de separadas, a replicação de uma das fitas (leading - líder) se dá de forma contínua no sentido do movimento da forquilha de replicação 5’→3’, e da outra fita (lagging) de forma descontínua e em direção oposta ao movimento da forquilha, formando os Fragmentos de Okazaki. Para completar os segmentos da fita descontínua, a DNA ligase une os fragmentos de Okazaki fazendo ligação fosfodiéster e formando uma fita contínua. A DNA polimerase III exerce um papel primário na catálise de nucleotídeos para formar a nova fita de DNA . A replicação do DNA é o processo responsável pela duplicação do material genética durante a divisão celular e formação de novas células (passagem da informação genética de célula a célula) associadas ao desenvolvimento do indivíduo, bem como no processo de reprodução (passagem da informação genética de geração a geração).
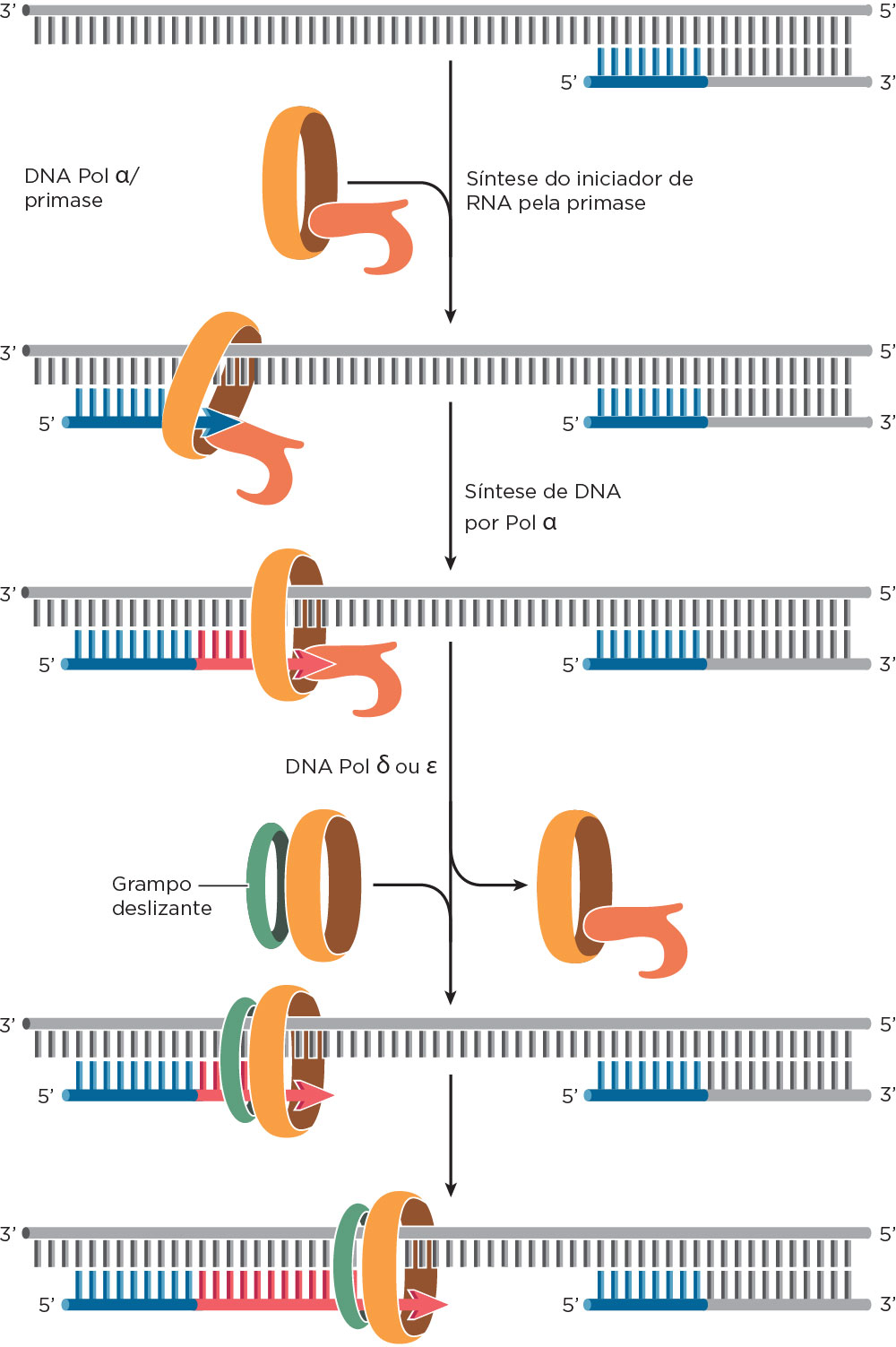
As células eucarióticas apresentam várias DNA Polimerases, sendo três dessas polimerases com suas subunidades essenciais à replicação, são estas a DNA Polimerase α/primase, DNA Polimerase δ e DNA Polimerase ε. No que se refere às funções, a DNA Polimerase α/primase, com quatro subunidades, está envolvida ao início do processo de replicação para formação das novas fitas por meio da síntese do primer iniciador (duas subunidades primase) seguida do início da polimerização de nucleotídeos pelas duas subunidades α. As DNA Polimerases δ e ε substituem a DNA Polimerase α aumentando a velocidade de síntese, por apresentarem alta processividade. Esse processo de troca de polimerases é chamado de polymerase switching atuando na origem de replicação de eucariotos. Como em procariotos, outras DNA polimerases também têm função envolvida no reparo do DNA que apresentam bases incorporadas erroneamente causando distorção na molécula. Essas atividades podem ser de exonuclease 5’→ 3’ ou 3’→5’. O aumento na processidade na replicação pelas DNAs Polimerases é devido ao ancoramento a outras proteínas que formam um grampo/anel deslizante sobre a fita de DNA, circundando-a e permitindo que a DNA Polimerase faça a síntese da nova fita de maneira mais eficiente .
A maquinaria replicacional não consegue replicar completamente os cromossomos lineares de eucariotos, os quais apresentam regiões específicas, tais como, os centrômeros e os telômeros, importantes no processo de divisão celular e na estabilidade cromossômica . Os telômeros se localizam na porção terminal dos cromossomos e apresentam uma composição de bases com sequências repetitivas em tandem (sequencial) de aproximadamente 10 à 15Kb (1 Kb corresponde a 1000 pares de bases – pb), variável entre os diferentes organismos, rica em GT. Como exemplo, em humanos a sequência telomérica é (5’-TTAGGG-3’)n.
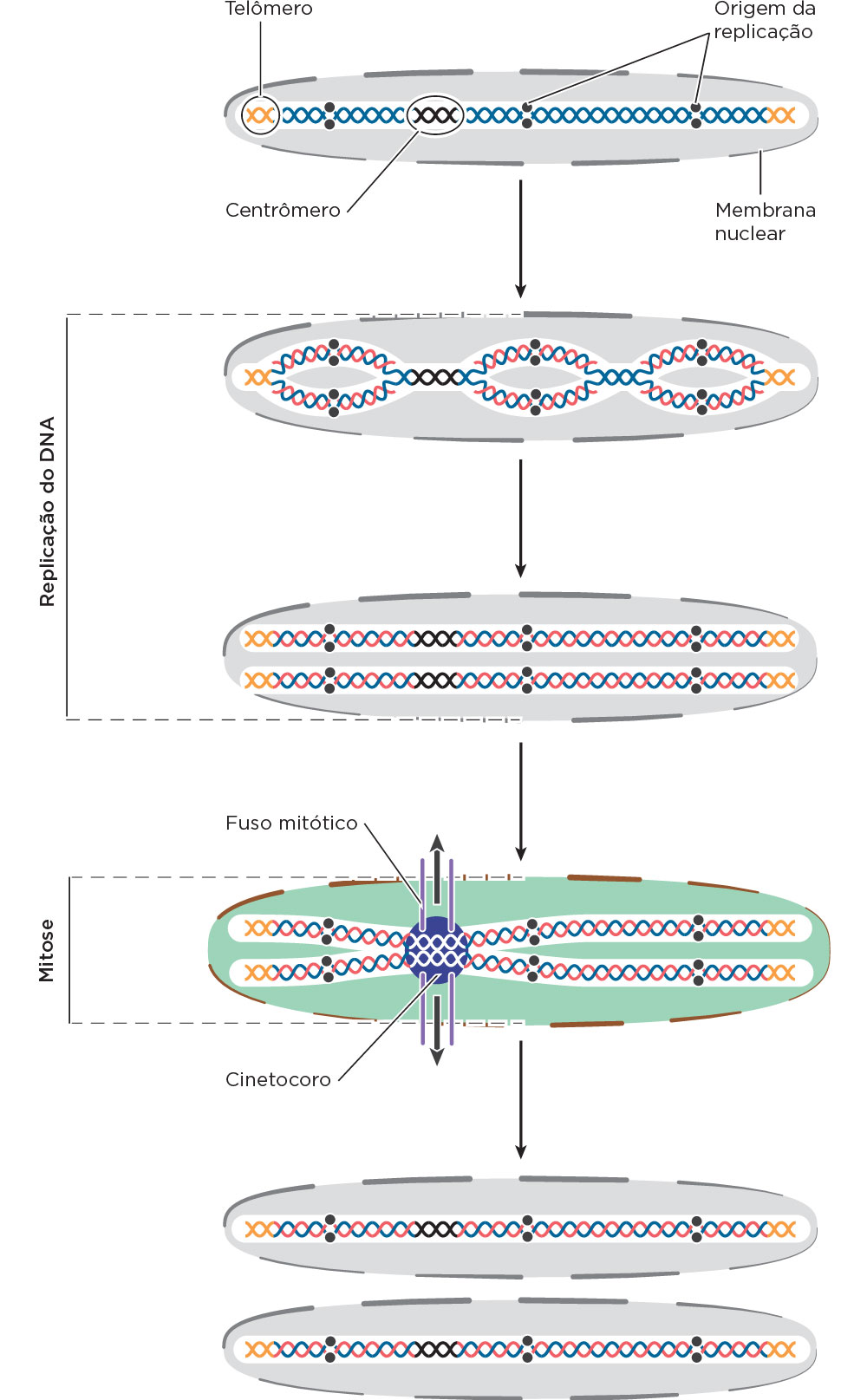
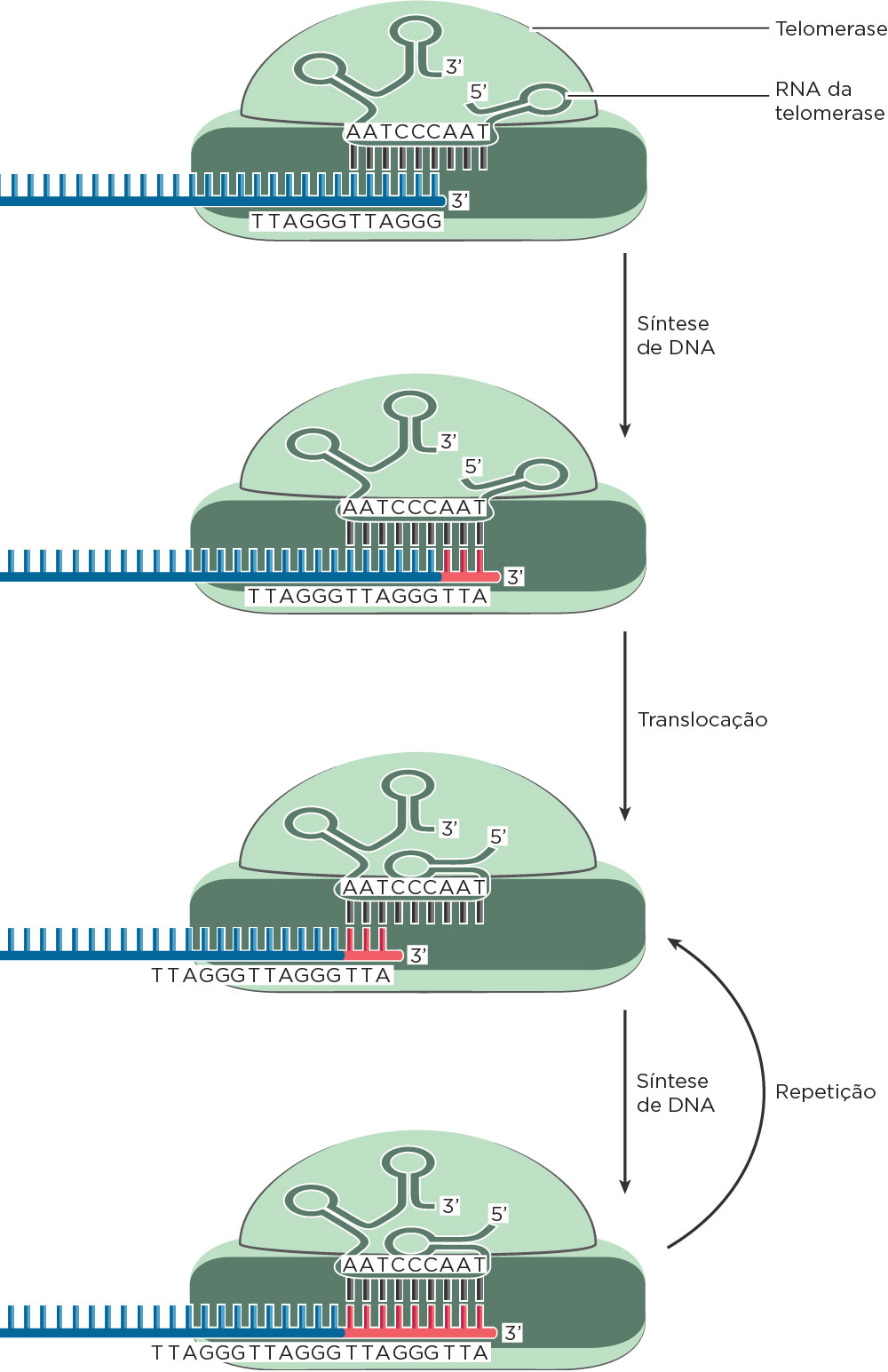
A falta de síntese de DNA das pontas na fita tardia ou descontínua a cada ciclo de replicação acarreta a perda de algumas unidades de repetições que formam os telômeros. Em processos de várias divisões celulares seria esperado a perda progressiva dos segmentos terminais da fita de DNA o que poderia atingir sequências codificadoras, levando à perda de informações genéticas essenciais à célula. O comprimento do telômero está associado à contribuição no envelhecimento celular normal. Algumas células, no entanto, exibem a ação da enzima telomerase de eucariotos; uma enzima com ação de transcriptase reversa diferenciada, que contém uma pequena molécula de RNA, que serve de molde para a extensão dos telômeros se ligando ao ssDNA . Essa atividade tem sido observada como sendo necessária para a imortalização celular no crescimento tumoral, com atividade altamente regulada durante a oncogenese, assim como no desenvolvimento. Nesse sentido, a modulação da ação da enzima telomerase pode ter implicações nas terapias anticancer e antienvelhecimento.
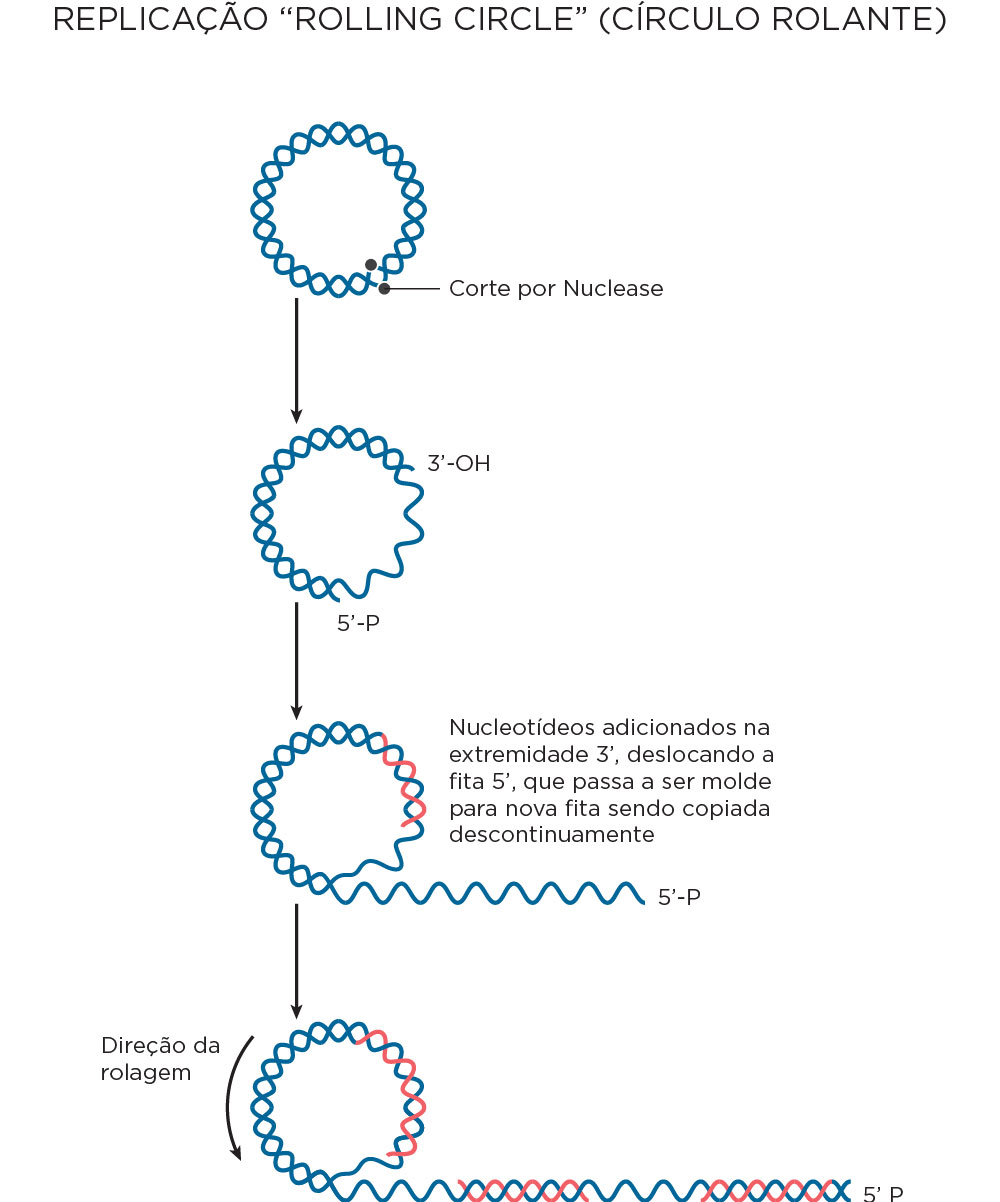
Outro modelo perculiar de replicação é a chamada círculo rolante, utilizada por algumas moléculas circulares de DNA como os plasmídios e em alguns tipos de vírus. Nesse modelo de replicação, o DNA circular parece girar à medida que desenrola um filamento contínuo pela ação de endonucleases. O corte feito por essas enzimas em uma das fitas de DNA fornece uma extremidade 3’-OH para ação da DNA Polimerase. À medida que a fita nova vai aumentando em tamanho, a molécula de DNA vai girando .