Unidade VI - Regulação da expressão gênica em eucariotos
3. Regulação da expressão gênica em nível pós-transcricional
Dentro os mecanismos de regulação em nível de pós-transcrição estão o processamento do RNAhn, necessário para que a molécula de RNAm final seja funcional, a recomposição alternativa que ocorre em muitos tecidos para atender as diferentes necessidades dos tecidos para as proteínas, já estudados na Unidade III. Dentre outros eventos que ocorrem em nível de regulação pós-transcricional estão a edição do RNA (RNA editing) e o silenciamento gênico pós-transcricional por RNAs de interferência.
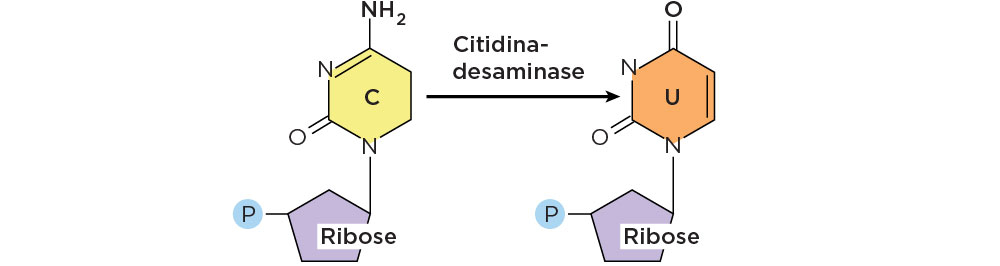
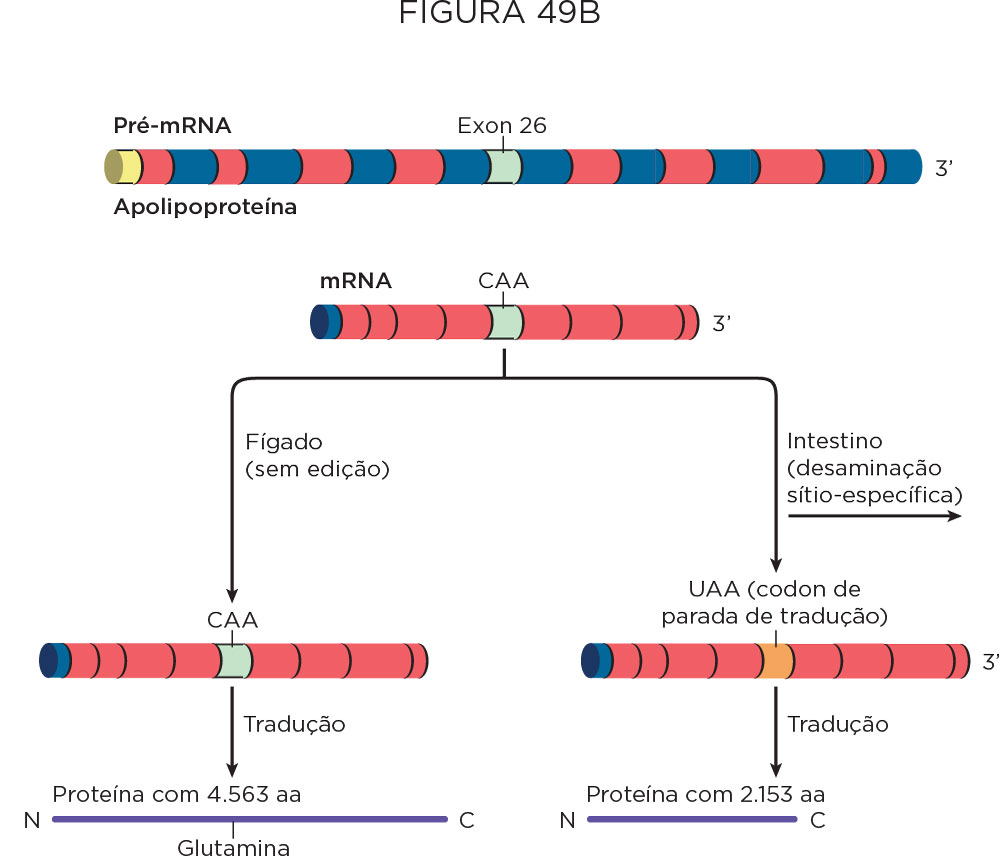
A edição do RNA (RNA editing), assim como o splicing alternativo, é importante na produção de proteínas que exercem função tecido-específica para a produção de diferentes isoformas a fim de atender a necessidade tecidual. No entanto, essas isoformas são produzidas pela atuação de uma enzima que atua na edição do RNAm maduro alterando codons na molécula de RNA. Um exemplo pode ser dado para as proteínas de baixa densidade como a apolipoproteína (apo), as quais são requeridas pelo fígado e pelo intestino, ambas envolvidas no metabolismo de lipídeos. O RNAm no fígado codifica para a proteína apoB-100 (sem edição), envolvida no transporte do colesterol e dos triglicerídeos produzidos por síntese endógena. No intestino, esse RNAm é editado pela ação da enzima citidina-desaminase, que converte a base “C” em “U" levando à formação de um códon de parada no meio do RNA e a proteína formada é uma apoB-48 (mais curta), envolvida no transporte dos lipídeos provenientes da dieta para os diversos tecidos. A desaminação ocorre assim, de modo tecido-específico e a mensagem é editada nas células intestinais, mas não nas células hepáticas. Como produto final de tradução, o códon CAA no fígado é traduzido como glutamina e no intestino os nucleotídeos “C" sofre desaminação, gerando um códon de térmmino de tradução UAA .
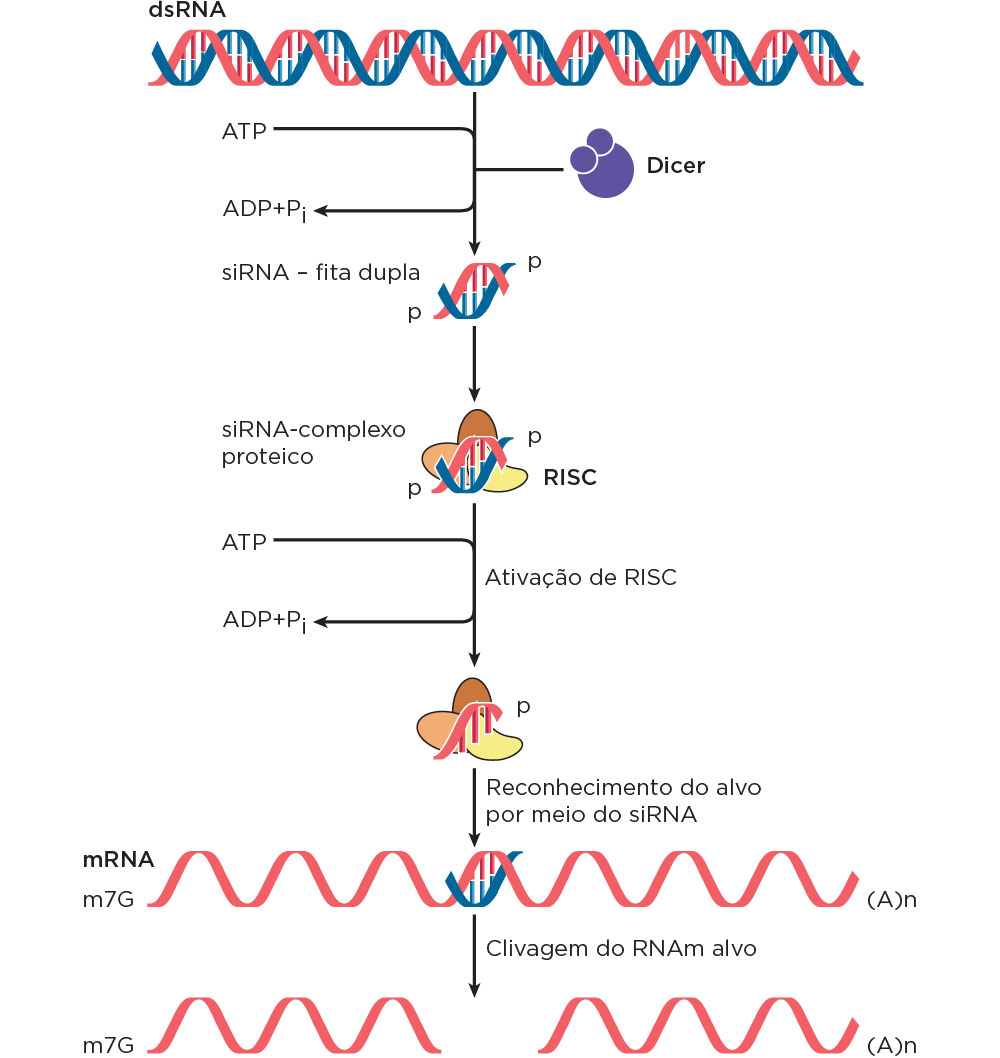
Os silenciadores gênicos pós-transcricionais (PTGS) constituem numa molécula de RNA que apresenta homologia com outra molécula de RNA, transcrita por um determinado gene. Assim, quando a molécula de RNA é transcrita, a outra molécula homóloga se liga ao RNA, impedindo que o gene se expresse. Em experimentos realizados com o nematódeo C. elegans, pelos pesquisadores norte-americanos Andrew Fire e Craig Mello, contemplados com o Nobel de medicina/fisiologia em 2006, foi observado que a introdução de RNAds (fita dupla; abreviação de double-stranded) na célula reduzia a expressão de genes que continham sequências idênticas (ou muito semelhantes) a uma das fitas desse RNAds. Esse mecanismo ficou conhecido como regulação da expressão gênica por RNAs de interferência (RNAi) e os detalhes envolvidos nessa regulação foram posteriormente descobertos. Os RNAi endógenos, ou seja, produzidos pela própria célula foram chamadas de miRNAs (microRNAs) para diferenciar dos RNAi que têm o mesmo efeito supressor de expressão gênica, no entanto, de origem exógena, chamados de siRNAs (short interfering RNA).
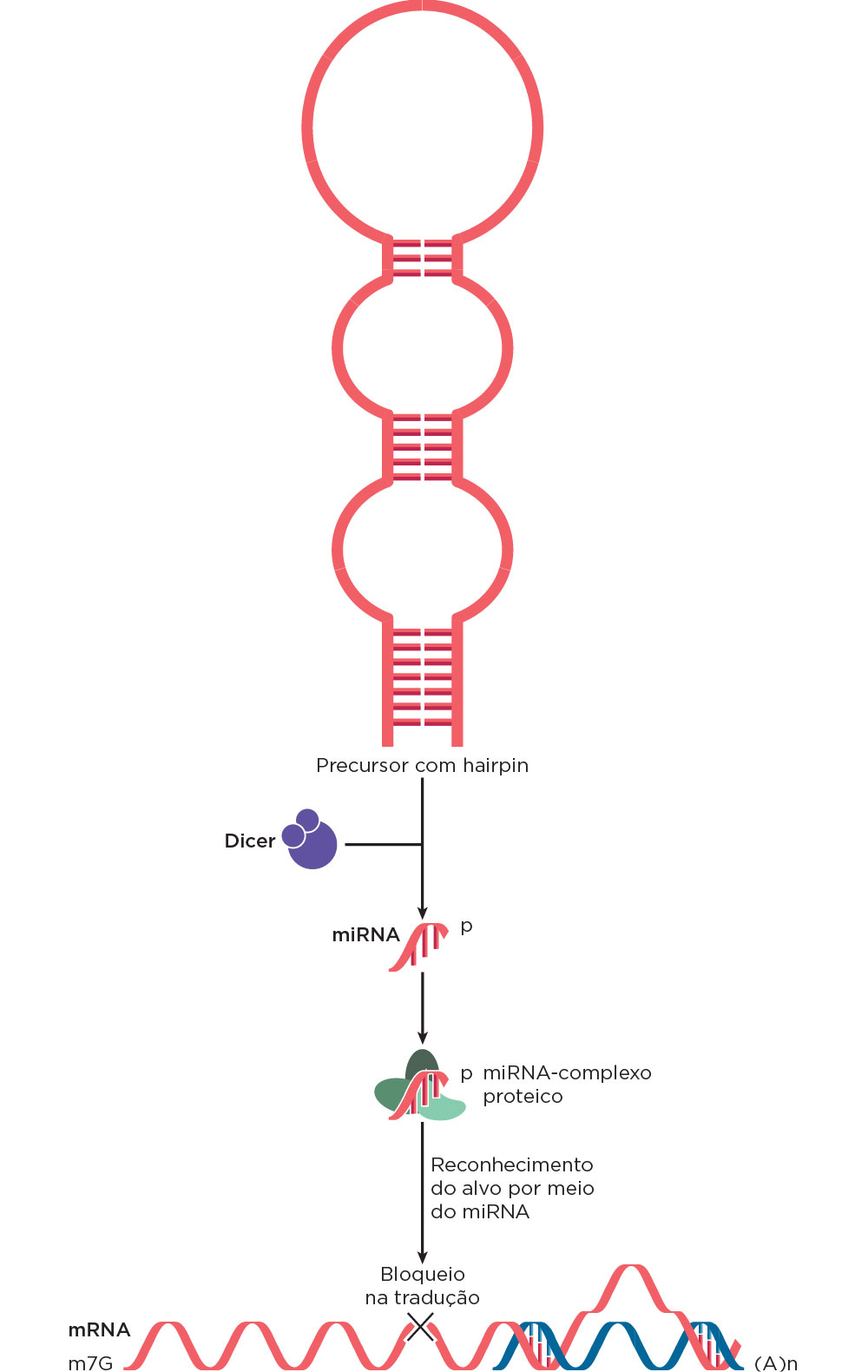
Os siRNAs provenientes de origem exógena (vírus ou artificial) apresentam um completo pareamento com o RNAm alvo frequentemente em apenas um sítio, enquanto os miRNAs são formados de um gene próprio a partir de exon, intron ou de síntese de RNA de regiões intergênicas e apresentam um pareamento incompleto podendo ser complementar a múltiplos sítios de um alvo. No núcleo, os precursores de miRNAs são longos, com cerca de 70 a 90 nucleotídeos de comprimento, e após serem processados em precursores menores (pré-miRNA) pela enzima Drosha (função de RNase) são transportados para o citoplasma pela proteína exportina. Os siRNAs não sofrem esse transporte. Os siRNAs não têm sido encontrado naturalmente em mamíferos, contudo a molécula relacionada, miRNA, tem sido amplamente descrita em vários organismos e tipos celulares.
No citoplasma, as moléculas de dsRNA são reconhecidas por uma enzima com função de RNase, a Dicer, a qual produz os siRNAs/miRNAs. Os RNAi clivados apresentam 21-23 nucleotídeos de dsRNA. Esses RNAi são incorporados em um complexo multi-protéico, o RISC (complexo de silenciamento gênico induzido por RNA), levando ao rompimento da dupla fita do dsRNA e uma das fitas é guiada pelo RISC a seu alvo de RNA, que apresenta homologia para clivagem endonucleolítica ou impedimento da passagem dos ribossomos durante o processo de tradução.
O que podemos observar aqui, é que, embora as proteínas sejam efetoras fundamentais da função celular, a base da complexidade e variação fenotípica pode primariamente ser devido a um sistema paralelo de informação necessária para a coordenação e modulação da expressão gênica por RNAs. Dentre dessa complexidade de RNAs vistos até agora estão os RNAs não codificantes (RNAnc) funcionais, como os RNAr, RNAt, snRNAs, ribozimas, miRNAs e siRNAs. Em humanos, aproximadamente 98% de todos os produtos transcritos correspondem a RNAnc.
O mecanismo da RNAi pode ter evoluído para proteger as células de elementos infecciosos ou desestabilizadores de genes, como certos vírus e transposons, que utilizam em seu sistema de replicação um dsRNA intermediário. Alguns vírus de ambos, plantas e animais, possuem um genoma de dsRNA. Em muitos outros vírus seus genomas são de RNA, que ao entrar na célula hospedeira é rapidamente convertida em dsRNA. Então o RNAi pode ser uma arma poderosa para conter a infecção por estes vírus através da destruição dos RNAm e assim bloquear a síntese de proteínas virais essenciais. Os transposons consistem em um tipo de DNA localizado no genoma de organismos complexos, com propriedades de saída do DNA e reinserção em outro local, podendo causar danos se eles se inserem em genes ou sequências regulatórias importantes. Os retrotransposons tem seu DNA copiado em RNA, os quais são transcritos reversamente em DNA e parte das moléculas dsRNAs são, assim, alvo de interferência pelas enzimas DICER e RISC, e os RNAi gerando podem destruir estes transposons impedindo sua inserção no genoma que pode provocar disruptura em genes essenciais .